文章信息
论文标题:Identification of a Selective Polymerase Enables Detection of N6-methyladenosine in RNA.
刊登日期:2013年12月
发表杂志:Journal of the American Chemistry Society(美国化学协会杂志)
影响因子:14.375
研究机构:斯坦福大学化学系Kool课题组

对于想要具体对某一个m6A甲基化位置进行精确定位的老师,目前常用的方法是m6A-CLIP-seq从全基因组层面进行鉴定。另外,对于m6A甲基化程度较高的基因,绝大部分课题组采用MeRIP-seq的方法先初步确定基因的某个区域,然后再采用MeRIP-qPCR的方法进行后期验证。这里我们将要介绍一种低通量验证具体m6A甲基化位点的方法。来自斯坦福大学化学系的Kool教授团队,在2013年成功从极端嗜热菌Thermus thermophilus中鉴定出一种逆转录聚合酶。这种特殊的聚合酶,针对未发生m6A修饰互补配对的T碱基具有高达18倍的选择性。换句话说,这种聚合酶一旦发现具有m6A修饰的腺嘌呤A后,逆转录扩增将会停止。Kool教授认为,这种酶通过分析电泳条带可以对RNA中m6A具体修饰位置进行定位及定量分析。此外这种酶与非特异选择性聚合酶(nonselective polymerase)在性能上进行了比较,分析两者在验证高丰度m6A修饰RNA的区别。最后Kool教授在28s rRNA上的第4190位精确定位到了m6A修饰。
鉴定m6A修饰的功能是一项艰巨的任务。早期不少实验室采用酶消化和同位素放射性标记的方法来鉴定m6A所在的RAC motif。这些研究还表明,在任何位点的m6A甲基化水平都是不太完整的,通常结果也只能说明RNA某个位点m6A修饰水平在22-99%之间,无法做到100%。
高通量测序的技术进步与抗体特异性富集等方法相结合后,不少课题组对传统的RIP-seq进行了针对m6A课题研究的改良和升级——MeRIP-seq。这种方法的优势在于可以几乎从全基因组层面鉴定所有的mRNA、lncRNA和circRNA等RNA上的m6A修饰情况。不过这种方法通常只能对高甲基化区域进行鉴定,而不能具体到某个m6A位点。
在此基础上,miCLIP-seq以及CLIP的升级版enhanced CLIP由此诞生。前者会涉及到同位素标记,而后者在牺牲部分精度情况上能够大大提升实验的通量且无需同位素标记。
然而,上面提到的所有方法都需要在酶消化的帮助下才能进行。若没有酶的消化,会出现腺嘌呤上的碱基修饰情况仍然精度不够等缺点。所以判断RNA在哪个碱基处断裂(RNA长度不同导致后期电泳长度不同)无疑是一种几乎不需要借助酶消化就能达到目的的方法,这时候,咱们提到的聚合酶就要登场了。作者推测极有可能存在一种只对m6A具有显著特异选择性的聚合酶,并且这种酶可能被用于位点特异性修饰检测。
作者终于在一大堆细菌的candidate聚合酶中,找到了这种特殊的聚合酶。这种特异性逆转录聚合酶取自一种极端嗜热菌Thermus thermophilus中。聚合酶因其对碱基的空间结构极其敏感,所以是一种新型的碱基修饰定位验证机制。
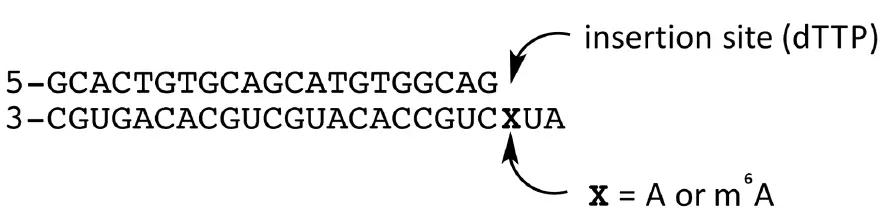
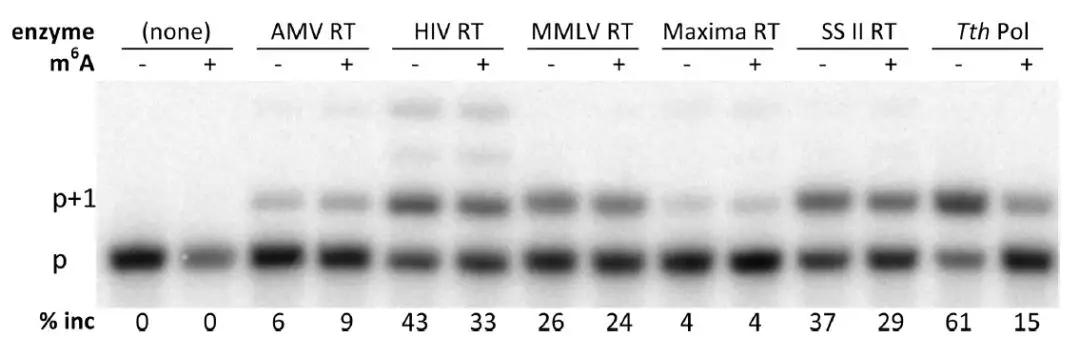
那么他们究竟是怎么做的呢?首先作者对大量具有逆转录酶活性的candidate进行第一步筛选。RNA模板设置有两组,一组带m6A修饰一组不带m6A修饰,以此作为底物模板来监测这些酶是否对被同位素标记的三磷酸胸苷(dTTP)DNA引物有延伸能力。在诸多的逆转录酶中,嗜热菌Tth在所有参与测试的酶中表现出较强的选择性,并在Mn2+存在的情况下充当逆转录酶的角色。
由于Tth酶有在某个特定条件下对一个特定的RNA模板显示出极强的特异选择性,所以作者进一步测试了不同温度、反应时间长短以及buffer成分是否可能会增强其特异选择性。已知Mn2+能够降低酶选择性,然而作者却发现Mn2+对于Tth酶的逆转录聚合酶活性至关重要。
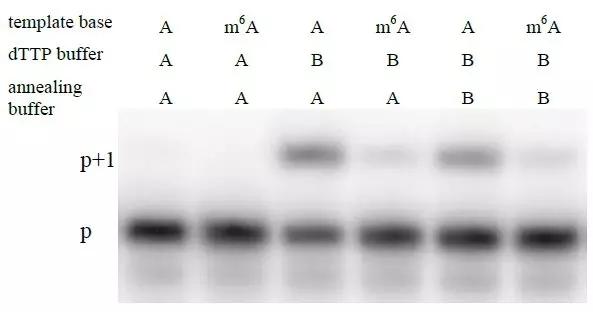
下一步,作者想要验证这种酶是否对其他序列具有延伸性。作者将小鼠的eef2基因的3’ UTR以及其他基因序列作为模板底物,这个模板分为带m6A修饰和不带m6A修饰的两份,并进行酶活性动力学验证。
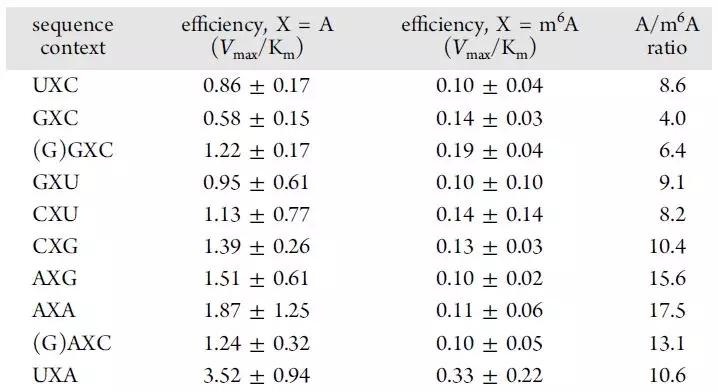
针对有m6A修饰的序列模板,在不含m6A修饰的对照组中Tth酶具有4-18倍的效率。整体上,不同背景下选择性差异可以忽略不计。例如T互补的A碱基在未发生修饰情况下,UAA序列相比其他序列具有更高的效率。此外,5’ G和3’ C都出现酶效率降低的情况。最值得注意的是,针对GAC motif,Tth聚合酶的活性提高了4-6.4倍。因此,这些现象均表明酶的选择性识别m6A修饰的motif是一个十分普遍的现象。
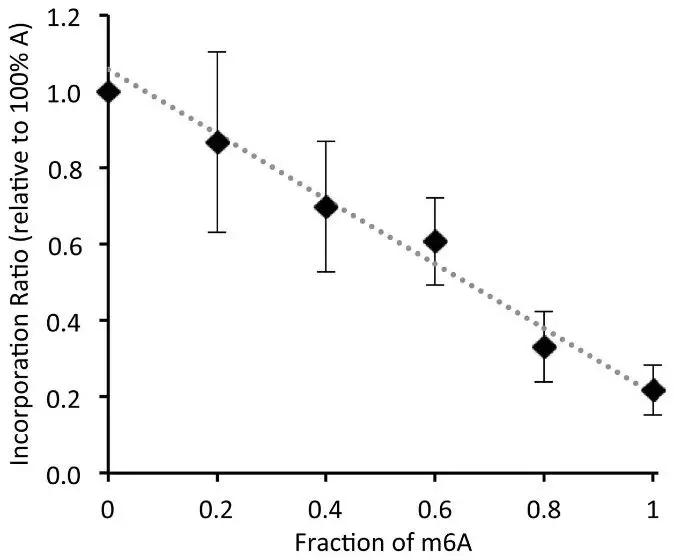
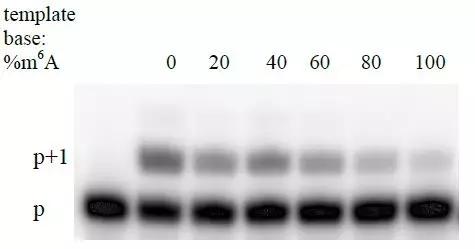
接下来作者要验证Tth酶可否对具体某个A碱基的m6A修饰程度进行定量。作者混入了已知m6A比例的RNA作为模板,在一个固定的时间点对dTTP消耗量进行测量。在已知的这份RNA模板中引物延伸的比例呈现出线性关系,这表明Tth酶可用于对具体的某个m6A甲基化位点进行定量。
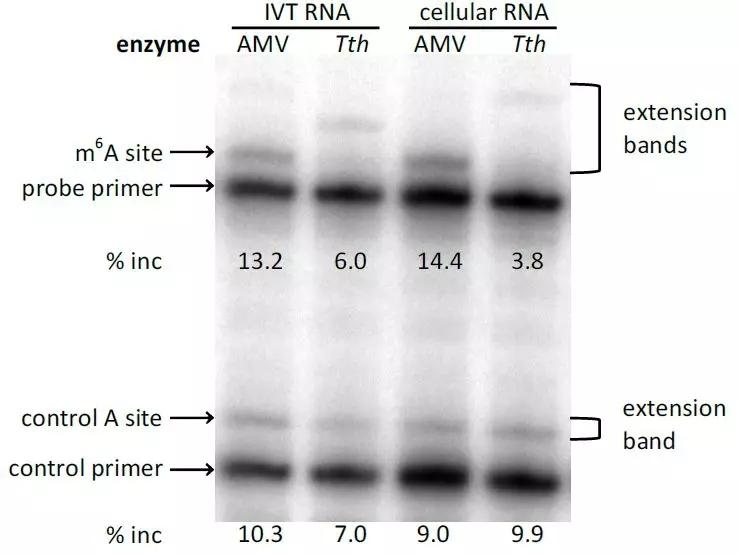
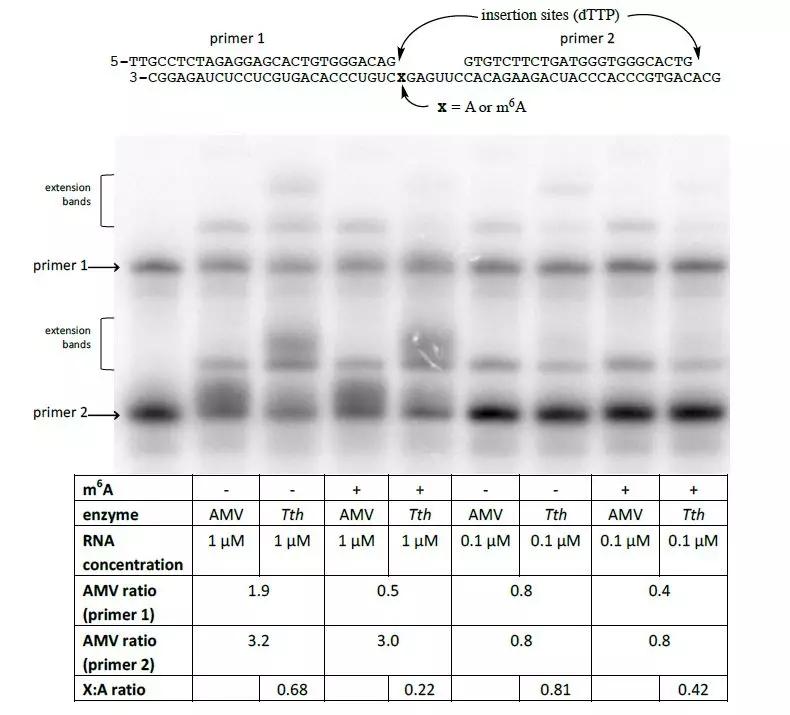
作者进一步验证Tth酶可否用于检测从哺乳动物细胞提取RNA中的m6A甲基化水平。由于RNA本身的浓度及其二级结构可能影响引物的延伸效率,作者引入了两种控制实验反应的方法。其中第一个策略是使用两种可以同时反应的引物(一个紧挨着被探测的甲基化位点,一个与已知的甲基化状态的碱基相邻)。这两种引物能够对样本中已知的RNA量进行测量。第二个策略使用的是非特异性聚合酶(即AMV RT逆转录酶)与同一种引物进行反应。之所以使用AMV RT逆转录酶是因为,在不同比例的RNA浓度下,AMV RT酶对Tth酶呈现线性相关。在感兴趣的碱基位点插入T后可用于检测非特异性酶AMV RT与Tth酶之间的区别。
已知人的核糖体RNA(rRNA)上的A碱基含有m6A修饰,包括18s rRNA的1832位,28s rRNA上的4189和4190位。针对邻近未发生碱基修饰进行酶消化后的比例,上面所述的几个rRNA的位点几乎存在100%的m6A修饰。作者针对这三个发生m6A修饰的位点设计了引物。从293细胞中提取total RNA,并对引物覆盖到的RNA位点进行标记。
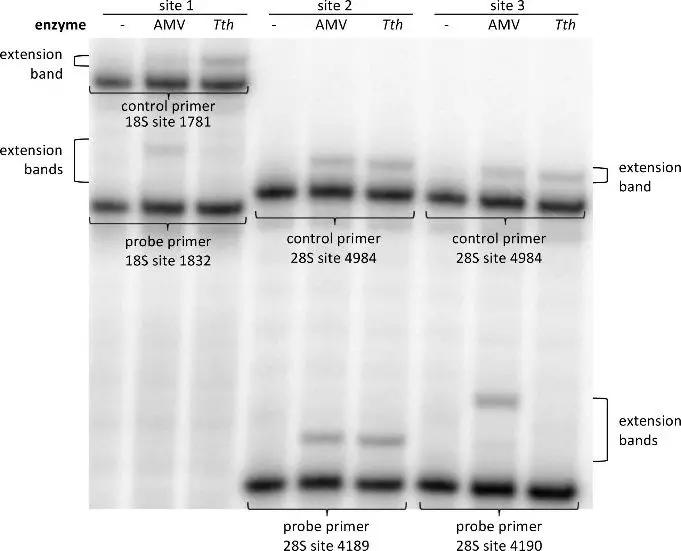
如上图显示,与对照组相比Tth组rRNA中1832和4190这两个位点对T的亲和度降低20%,并且针对这个作者使用人工合成的RNA进行了定量验证。与之相反的是,作者观察到相对于AMV RT逆转录酶,Tth酶对rRNA中4189位点的亲和度比对照组高出120%。因此作者认为未发生甲基化的位点就是非常明确地位于28s rRNA的4190而不是4189附近。
最后,作者试图在哺乳动物细胞中提取的mRNA中验证m6A修饰的具体位点。下一步作者选择bRPL基因3’ UTR序列上已知的m6A位点。已知bRPL的3’ UTR上m6A修饰水平在20%左右,作者在293细胞中过表达bRPL的3’ UTR序列。接下来设计两对特异性引物,检测已知的修饰位点以及腺嘌呤附近的碱基。
当RNA上含有m6A修饰的碱基位点时,亲和性显著低于不带任何m6A修饰的碱基。作为额外对照组,作者在体外表达了一个不带m6A修饰的bRPL,检测引物对其的亲和度。前期浓度较高的时候实际上,无论是细胞中提取的total RNA还是体外转录的RNA基本没有差异。然而在酶的浓度比较低时,m6A就发生了比较大的差异。这就说明293细胞中确实存在m6A修饰。
