单细胞多组学技术被Nature Methods评为2019年年度技术方法,紧接着空间转录组就被Nature Methods评为2020年年度技术方法,单细胞转录组测序加空间转录组联合分析已经是目前炙手可热的转录组研究方法,是研究细胞异质性、组织形态学、组织发育机制和细胞图谱等领域的不二选择。
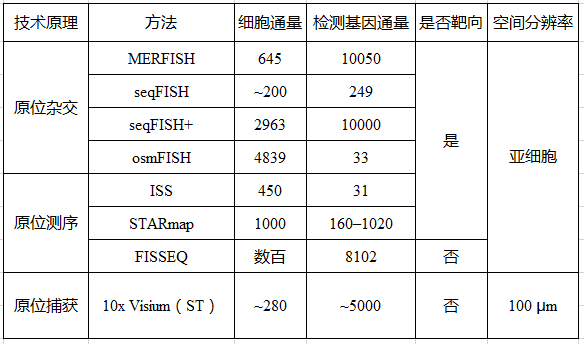
目前广泛应用的空间转录组技术10x visium(收购了Spatial transcriptomics技术),原理为原位捕获(in situ capturing),其不足之处是芯片一个位点上有1~10个细胞,无法做到单细胞级分辨率,且无法检测低表达量基因。MERFISH(multiplexed error-robust fluorescence in situ hybridization,多路抗错荧光原位杂交)发明者庄小威在Nature Methods发表了Spatially resolved single-cell genomics and transcriptomics by imaging一文,介绍了基于成像原理实现单细胞空间分辨转录组学的两类技术(表 1),这些技术能将RNA定位在亚细胞位置。
荧光原位杂交(Fluorescence in situ hybridization, FISH)应用荧光标记探针定位核酸分子,单分子水平FISH(smFISH)能够高度精确定位RNA在细胞中的位置。然而,有限数量的颜色通道限制了FISH的通量,只能检测10~30种RNA。为解决这个问题,2013年庄小威团队开发了MERFISH,通过来自多轮杂交的信号组合来识别基因,每个基因都分配一个N位二进制条形码,每位的“1”或“0”值对应于该基因是否在一个特定轮的杂交中被检测到(图1a)。该二进制编码方案可以通过N轮杂交区分2N个基因(当在N轮杂交中使用C种颜色进行成像可达到2NC个基因)。现在MERFISH可以通过23轮杂交和3色成像,实现了在单细胞中对大于10,000个基因成像。
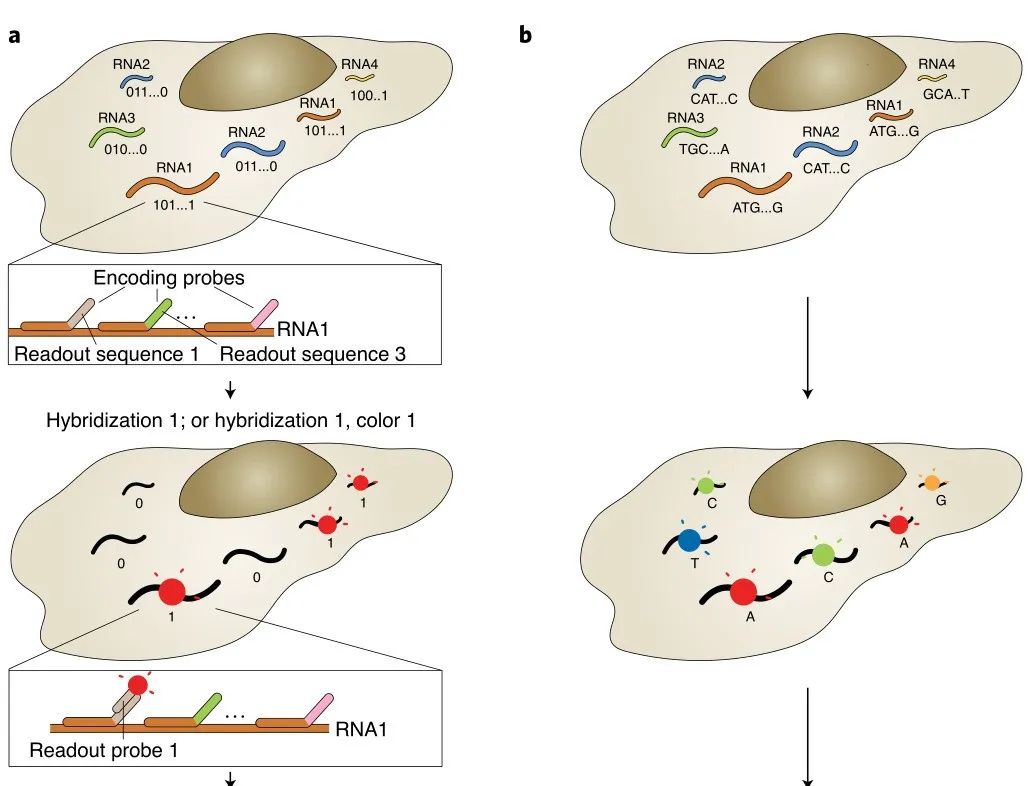
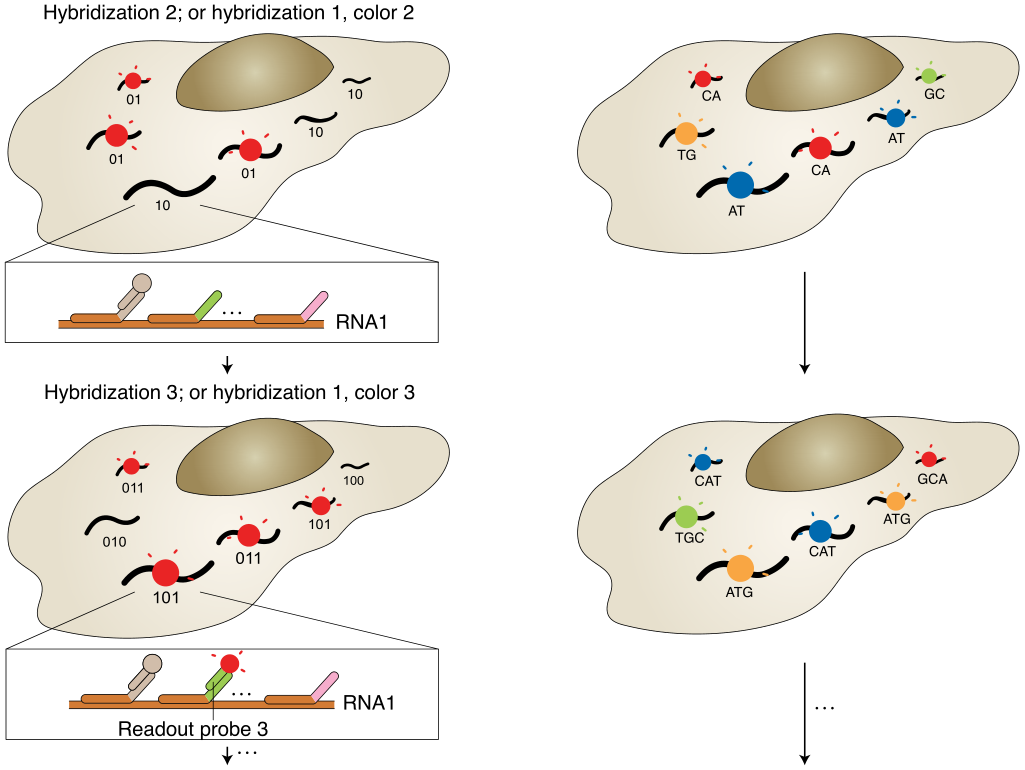
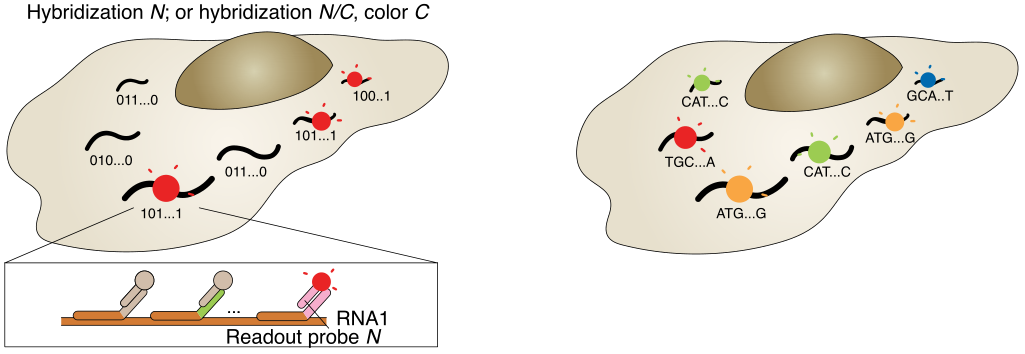
a,以MERFISH方法为例的多路FISH方案。本文展示了一种二进制条形码方案,以及条形码如何用编码探针库印迹在RNA上,并使用不同的读出探针逐位检测。b,原位测序方案示意图。核酸序列可以是基因的内源性序列,也可以是分配给基因的条形码序列,通过测序进行逐个核苷酸检测,圆圈的颜色可能表示单个核苷酸或相邻的二核苷酸对。
另一种多路FISH方法seqFISH,通过顺序颜色编码来区分RNA,在seqFISH中,不同的RNA组合在每一轮杂交中被分配不同的颜色,之后用一组新的颜色组合的FISH探针与细胞RNA杂交,通过C色和N轮杂交,可以区分CN不同的基因。最初seqFISH实现了单细胞中12个基因的定位,之后通过引入杂交链信号放大实现了约250个基因成像。然而,显微成像时分子颜色堆叠问题限制了seqFISH的通量发展。
空间分辨的单细胞分析也通过序列杂交实现,例如osmFISH在每个杂交的每个颜色通道中对应单个基因。这种方法在组织成像中提供了极好的信号质量,尽管其多路复用性水平较低,但提供了一种对表达水平过高或序列过短的基因进行成像的方法,它们通常无法进行单分子检测或条形码识别。这种方法也可以与上述方法相结合,可以覆盖多数的基因和难以进行单分子检测的基因。
总的来说,多路FISH方法提供了具有高空间分辨率的原位单细胞转录组学和3D基因组分析。这些方法的另一个优点是检测效率高。MERFISH已经证明了对超过10000个基因中的80%成像,如此高的检测效率可以定量分析低表达水平的基因。
与此同时,基于成像的原位测序方法也被开发出来,用于具有高空间分辨率的单细胞转录组分析,在细胞中对RNA进行原位测序,四种颜色荧光各代表一种核苷酸,根据荧光信号记录序列信息(图1b)。不同于原位杂交针对靶向RNA,原位测序能以靶向或非靶向的方式进行。
在最初原位测序(ISS)技术中,需在靶向原位测序中预选一组基因,将特异性核苷酸序列作为条形码通过挂锁探针杂交传递到这些基因,然后进行循环扩增和原位测序检测。STARmap使用条形码挂锁探针,与靶标杂交,通过添加第二个引物,针对挂锁探针旁边的位点,避免了逆转录(RT)步骤,并降低了噪音。STARmap能够对完整的组织样本进行3D分析,保留了细胞的方向性,而不仅仅是单一的2D层。但需要注意的是,这种能力只表现在100~150微米厚的切片和较少的目标数量上。STARmap能够对单细胞中1020个目标基因成像,检测效率与单细胞RNA测序相当。
在非靶向原位测序中,例如FISSEQ(Fluorescence in situ sequencing,荧光原位测序),引入了细胞RNA逆转录为cDNA后扩增,并无需任何预选靶向序列。这种方法的优点是能做到全基因组覆盖,FISSEQ已经在同一样本中对大于8000个基因成像。然而,由于所有种类的RNA都被测序,为了避免分子拥挤的问题,FISSEQ需要使用测序引物检测单个分子,导致只有一小部分的扩增子被测序,使得检测效率较低,为全基因组0.2%到<0.01%。
对于所有的靶向方法,包括原位测序和多路FISH,这是克服分子堆叠以及提高检测范围一种简单的方法——将目标基因分成多个组,一次一组成像。例如,10000或20000个基因可以分为10或20组1000基因(通常不超过10轮多色成像),对10000或20000基因成像不需要改变方法,只是进行多次10或20轮成像。
之后小编将带来基于图像的单细胞空间分辨和转录组学技术应用介绍,敬请期待!
1.Method of the Year 2019: Single-cell multimodal omics. Nat Methods. 2020;17(1):1. doi:10.1038/s41592-019-0703-5 2.Method of the Year 2020: spatially resolved transcriptomics. Nat Methods. 2021;18(1):1. doi:10.1038/s41592-020-01042-x 3.Ståhl PL, Salmén F, Vickovic S, et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics. Science. 2016;353(6294):78-82. doi:10.1126/science.aaf2403 4.Zhuang X. Spatially resolved single-cell genomics and transcriptomics by imaging. Nat Methods. 2021;18(1):18-22. doi:10.1038/s41592-020-01037-8 5.Liao J, Lu X, Shao X, Zhu L, Fan X. Uncovering an Organ's Molecular Architecture at Single-Cell Resolution by Spatially Resolved Transcriptomics. Trends Biotechnol. 2021;39(1):43-58. doi:10.1016/j.tibtech.2020.05.006
