云序生物针对环状DNA特异性的junction位点,采用了outward-facing引物设计策略,示意图举例如下:针对线性DNA,primer 1和primer 2是尾对尾,无法有效扩增;而如果线性DNA的1和3首尾相连形成了闭合的环状DNA, primer 1和2就可以扩增出针对特异性junction位点的产物,从而特异性的检测环状DNA
。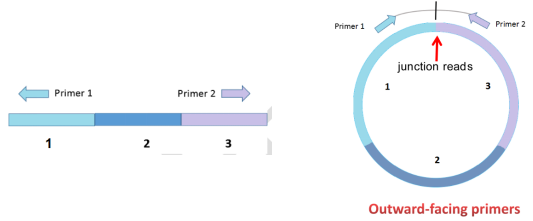
线性DNA引物设计(尾对尾) 环状DNA引物设计(首尾相连)
具体操作步骤
具体步骤主要分为三步,分别是碱基序列的获取、转换以及引物的设计。以染色体坐标为chr1:100,021,994-100,023,255的eccDNA为例,进行碱基序列的获取及引物设计。
一、获取碱基序列
1、打开UCSC genome browser 网站,点开Genomes栏,选择测序参考的基因组,如hg19。
UCSC genome browser 网站:http://genome.ucsc.edu/cgi-bin/hgGateway

图 1
2.选好基因组版本后,输入eccDNA 的位置坐标“chr1:100,021,994-100,023,255”,点 “GO”(注意:坐标的格式及符号需按模板要求)。
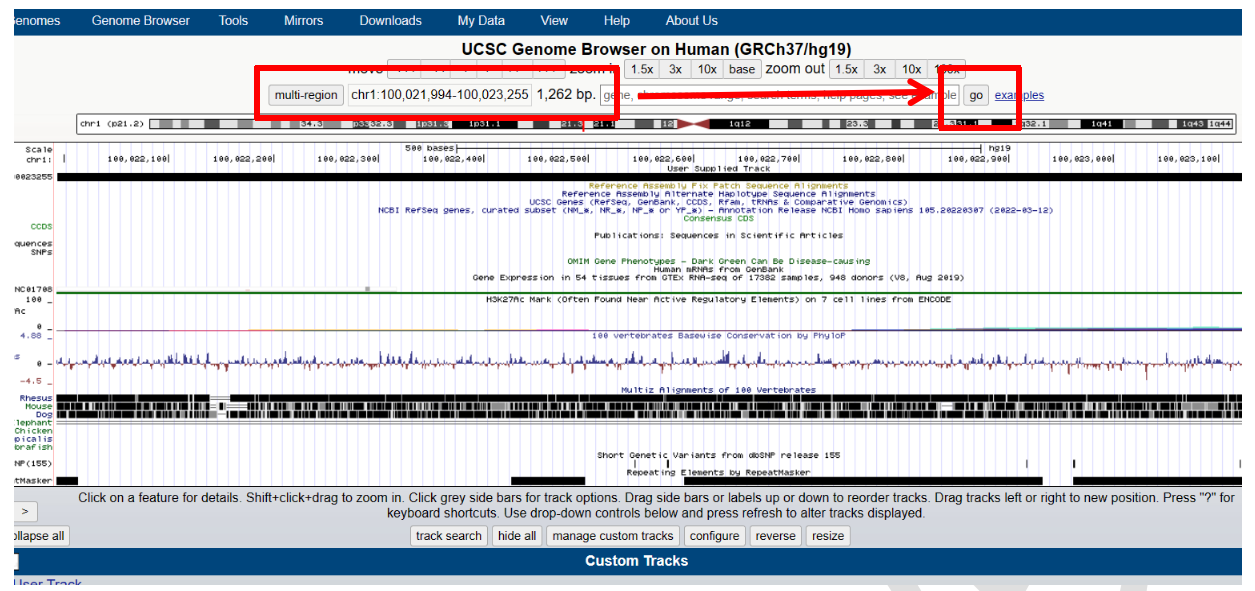
图 2
3. 进入如上图2 中chr1:100,021,994-100,023,255坐标序列的界面后,键盘按“ V+D”键,即可跳出如下图3的新界面(注:有时候按V+D键不一定立马弹跳出界面,需多次尝试);在跳出的新界面中点击“get DNA”,即可跳出对应坐标的eccDNA碱基序列信息(如图4所示)。
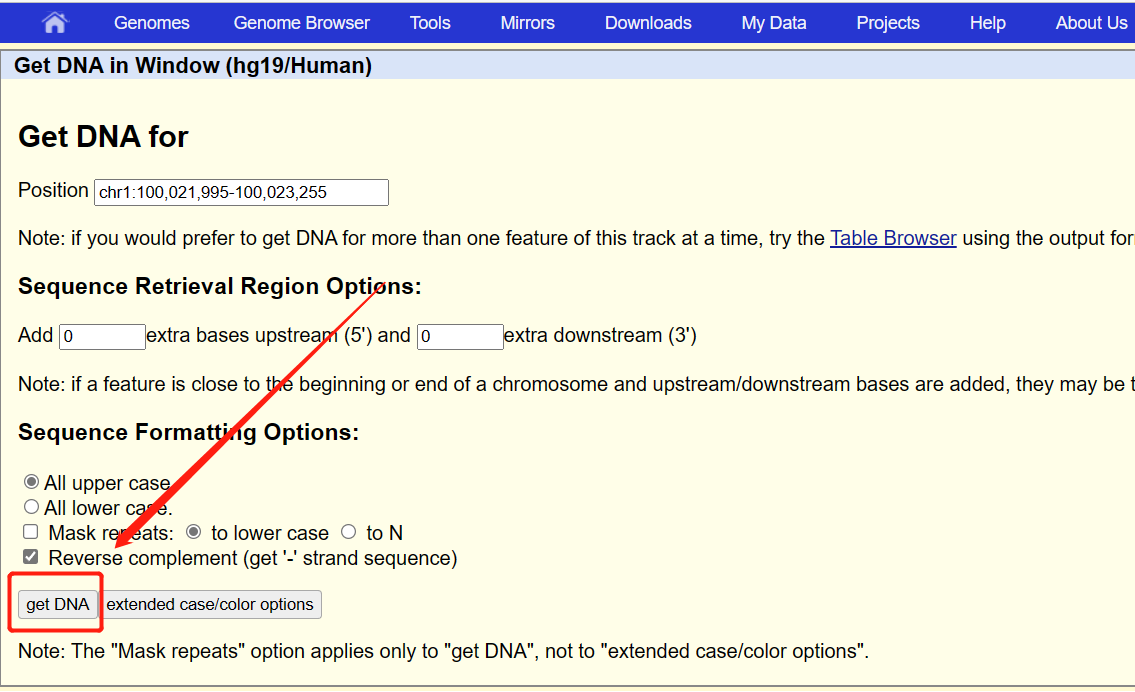
图 3

图 4
二、序列转换
1. 按以上步骤获取的序列为eccDNA对应的线性DNA序列,将首尾相连即为eccDNA的环状序列。将eccDNA序列的5’端300bp左右的序列用绿色高亮标记,3’端300bp左右的序列用黄色高亮标记,以chr1:100,021,994-100,023,255为例,TA为eccDNA的junction位点。

2. 将eccDNA全长序列的3’端300bp序列移至5’端300bp序列上游,得到如下序列:

3. 针对步骤5中的600bp序列设计对应的PCR引物,设计方法同常规引物设计即可,但forward和reverse两条引物需要分别位于蓝色区域和黄色区域(即TA的上游和下游),以确保扩增产物中包含eccDNA的junction 位点。
注:如eccDNA全长小于600bp,则可将300bp相应缩短,只需保证最终扩增产物中含有junction 位点,确定其成环状态即可。
三、设计引物
以primer5.0软件为例
1、打开 primer 5.0 软件,点击“File/New/DNA sequence“,如下图所示:

2、将上列转换后的600bp序列复制到软件中,如下图:
3、点击Primer按钮进行引物设计:

4、点击 search按钮,在弹出的以下对话框中修改引物参数:
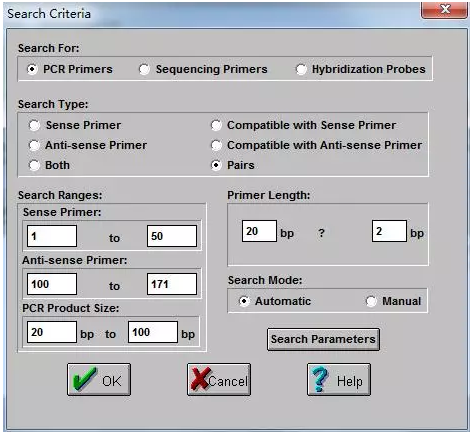
5、填写以上参数后,点击OK按钮,程序会弹出引物Search结果(如下图),点击OK,会弹出可选引物评分:
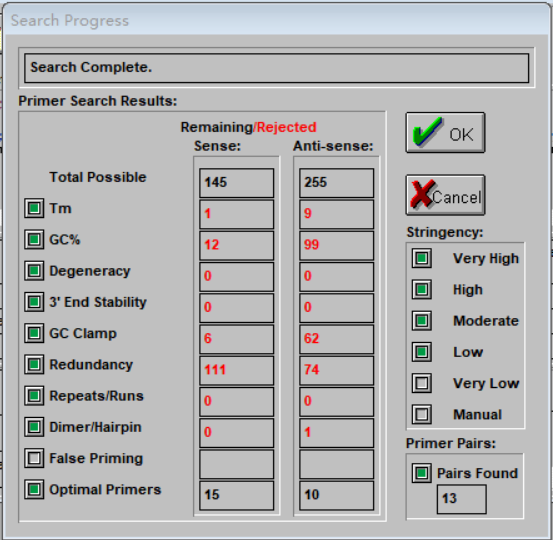
6、引物筛选:对软件给出的引物根据系统评分从高到低进行筛选。选取对应eccDNA评分最高的引物进行评估,如下图所示:
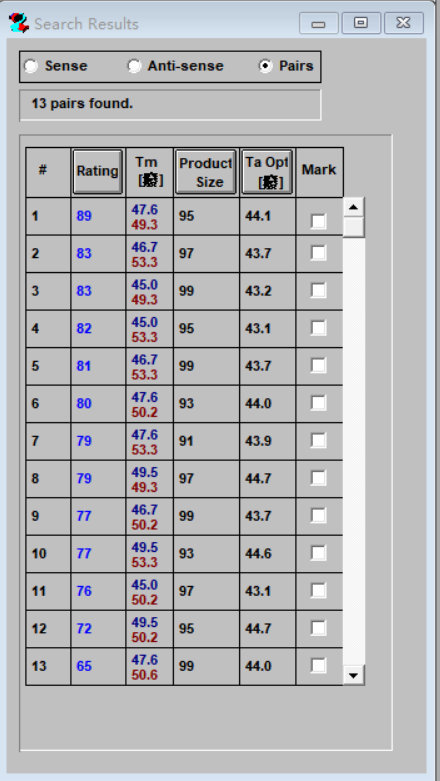
7、引物评估:使“Sense”中的“Tm”与“Anti-Sense”中的“Tm”差值的绝对值不大于2.5,“GC%”=40%~70%;核对“Hairpin”,“Dimer”,“False Priming”,“Cross Dimer”,调整后结果如下图所示:

8、点击“Edit”/“Copy”,输出“Sense primer”及“Anti-sense primer”,即得引物序列信息。
