变异检测
产品名称: 变异检测
英文名称: variant calling
产品编号:
产品价格: 询价
产品产地: null
品牌商标: null
更新时间: null
使用范围: null
- 联系人 :
- 地址 : 上海市浦东新区国际医学园区康新公路3399弄3号楼
- 邮编 : 201203
- 所在区域 : 上海
- 电话 : 159****9102 点击查看
- 传真 : 点击查看
- 邮箱 : marketing@majorbio.com
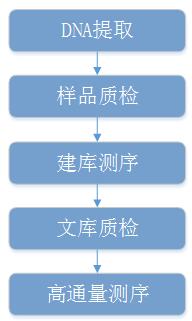
产品介绍
变异检测是指通过高通量测序技术对某一物种个体或群体的全部基因组进行测序及差异分析,获得大量的遗传变异信息,如单核苷酸多态性位点(SNP,Single Nucleotide Polymorphism)),插入缺失位点(InDel,Insertion/Deletion)及结构变异位点(SV,Structure Variation),拷贝数变异(CNV,Copy Number Variations)等分子标记。为后续的功能基因挖掘提供最基本和最全面的数据基础快速精准高效的解析基因组之间的差异,对全基因组的每一个碱基进行分析,获得最广泛的分子标记。
适用范围
有参考基因组的物种;
物种选取具有代表性。
技术优势
遗传变异类型丰富:单核苷酸多态性(SNP)、插入缺失(InDel)和结构变异(SV)一网打尽,深度解析基因变异的各个方面;
快速高效:仅仅45个工作日即可完成全部分析内容;
精准分析:10年以上项目经验的专业分析人员深入解析,精确解决科研过程中的各个问题。
技术流程
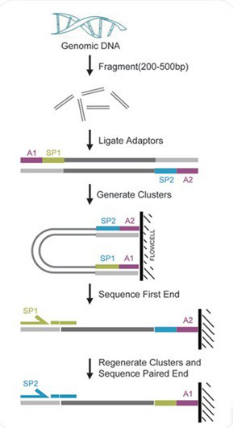
重测序建库及测序流程
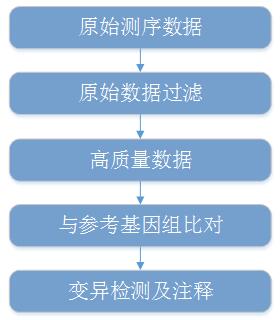
变异检测分析流程
|
分析内容 |
解决问题 |
核心软件 |
|
原始数据过滤 |
对原始数据质控,过滤低质量的序列 |
FastqQC/NGS-QC |
|
参考基因组比对 |
将测序的reads比对到参考基因组 |
BWA/samtools |
|
多态性检测 |
研究各类型变异及其在基因组上的分布 |
Samtools/GATK/CNVanotor/BreakDancer |
|
变异基因功能注释 |
统计变异基因分布规律 寻找变异丰富的功能分类 |
Gene Annotation (In house)/Anovar/SnpEff |
技术参数
产品
测序平台
测序策略
检测内容
项目周期
重测序
Illumina PE150
350bp文库
SNP、InDel、SV,CNV
45个工作日
变异类型
测序深度
SNP,small InDel
10×
SV
20×
CNV
30×
案例解析
产品
测序平台
测序策略
检测内容
项目周期
重测序
Illumina PE150
350bp文库
SNP、InDel、SV,CNV
45个工作日
变异类型
测序深度
SNP,small InDel
10×
SV
20×
CNV
30×
案例一、关键个体重测序检测家牛的基因组变异
对具有有代表性的关键个体进行全基因组重测序可以高效地识别群体的遗传变异。研究对代表了Fleckvieh品种69%的遗传多样性的43头牛重测序,测序平均深度7.46×,与参考基因组比对共发现了超过1,700万个变异位点,约90%为多态性位点(SNPs),10%为短插入缺失片段(InDels)。91,733个变异位于18,444个编码基因内,46%为非同义突变,其中575个变异预测会造成提前终止。对个体的低或中等深度重测序能够高效并可靠地识别群体中大部分的基因组变异,这些结果有助于后续基因功能研究,比如高精度的GWAS基因定位。
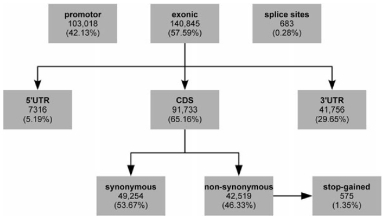
图1 变异的功能注释
案例二、个体重测序揭示甜高粱和普通高粱的基因组差异
高粱有普通高粱和甜高粱两个亚种,它们的许多重要的农业性状存在差异。文章对2株甜高粱和1株普通高粱进行了全基因组重测序,结果发现甜高粱和普通高粱有1,500个差异基因,这些基因与糖和淀粉的代谢,木质素和香豆素的合成,核酸代谢,压力反应和DNA损伤修复相关。共找到100万个SNP,9.9万个InDel、1.6万个结构变异和1.7万个CNV。主效的SNP,Indel以及大片段的插入和缺失大部分位于LRR(leucine rich repeats),PRR以及抗性基因上,生长相关的重要基因上的遗传突变却很少,图1。
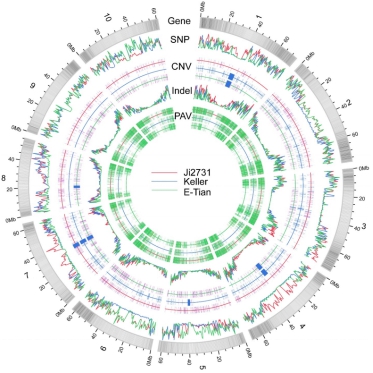
图1 高粱变异信息的可视化结果
参考文献
[1] Jansen S, Aigner B, Pausch H, et al. Assessment of the genomic variation in a cattle population by re-sequencing of key animals at low to medium coverage[J]. BMC Genomics, 2013, 14(1): 1.
[2]Zheng L Y, Guo X S, He B, et al. Genome-wide patterns of genetic variation in sweet and grain sorghum (Sorghum bicolor)[J]. Genome Biology, 2011, 12(11):287-302.
常见问题
(1) 想要研究某个物种的基因组学特征,是进行基因组de novo还是重测序?
答:如果您研究的物种有参考基因组,并且参考基因组的完整程度较好,建议您选择重测序产品,该产品的性价比更高。如果您的物种没有参考基因组或者参考基因组的完整程度较差,建议您选择基因组de novo。
(2) 一般可以用于变异检测参考基因组的mapping率要达到多少?
答:一般建议85%以上比较好,最低要求在65%以上,比对上的reads才会进行分析,太低时数据有效利用率降低。