SNP检测
产品名称: SNP检测
英文名称: single nucleotide polymorphism
产品编号: 1111
产品价格: 询价
产品产地: null
品牌商标: null
更新时间: 2024-06-12T09:46:24
使用范围: null
- 联系人 : 朱方伟
- 地址 : 闵行区瓶安路1358号久创科技园3号楼502
- 邮编 :
- 所在区域 : 上海
- 电话 : 152****1985 点击查看
- 传真 : 点击查看
- 邮箱 : market@jizhenbio.com
SNP介绍
SNP(single
nucleotide polymorphism),即单核苷酸多态性,是由于单个核苷酸改变而导致的核酸序列多态。一般来说,一个SNP位点只有两种等位基因,因此又叫双等位基因。
SNP绝大多数位于非编码区,多为转换,即一种嘧啶碱基换为另一种嘧啶碱基,或一种嘌呤碱基换为另一种嘌呤碱基,转换与颠换之比为2:1。SNP在CG序列上出现最为频繁,而且多是C→T,因为CG中C即胞嘧啶常被甲基化,自发脱氨后即变为胸腺嘧啶。
SNP在人类基因组中的发生频率比较高,大约平均每1000个碱基中就有一个多态位点。有些SNP位点还会影响基因的功能,导致生物性状改变甚至致病。单核苷酸多态性是继RFLP (限制性片段长度多态性)和小卫星DNA重复序列、微卫星DNA重复序列后的第三代DNA水平遗传多态性标记,是研究人类家族和动植物品系遗传变异的重要依据,因此被广泛用于群体遗传学研究(如生物的起源、进化及迁移等方面)和疾病相关基因的研究,在药物基因组学、诊断学和生物医学研究中起重要作用。
技术原理
提供各种SNP技术服务平台:FRET+基因分型平台,Taqman探针分型技术平台、Multiplex SNaPshot分型技术平台、LDR分型技术平台、飞行时间质谱(MALDI-TOF MS)分型技术平台、测序分型技术平台。涵盖了从低通量、中等通量到高通量的完整SNP分型平台,能满足不同规模项目的需求。
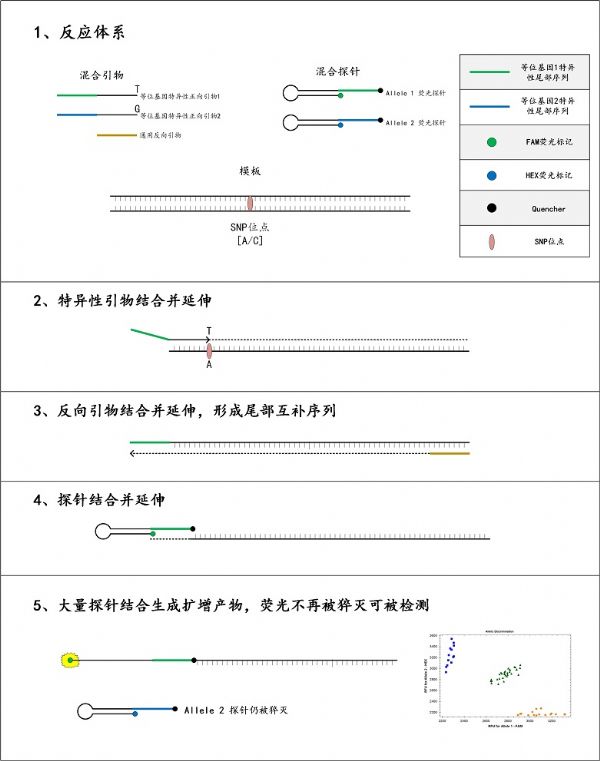
FRET+探针法
FRET(Fluorescent Resonance Energy Transfer),即荧光共振能量转移,是距离很近的两个荧光分子间产生的一种非放射性的能量转移现象。利用FRET+技术可在广泛的基因组DNA样本中(包括一些具有复杂基因组的DNA样本),对SNPs(single
nucleotide polymorphisms,单核苷酸多态性)和特定位点上的InDels(Insertions and Deletions,插入和缺失)进行准确的基因分型,该方法具有高度的稳定性和准确性。
FRET+基因分型技术特点
Ø优异的准确性和敏感性
Ø超强的灵活性
Ø超低的检测成本
Taqman探针法
PCR反应时,加入一对两端有不同荧光标记的MGB特异探针来识别不同等位基因,5’端为报告荧光基团(reporter),3’端为淬灭荧光基团(quencher)。PCR过程中,两个探针能与正向引物和反向引物之间的互补序列特异退火结合。当探针以完整形式存在时,由于能量共振转移,荧光基团只发出微弱荧光。特异的探针与相应的等位基因结合后,DNA聚合酶发挥5’到3’外切酶活性,把报告荧光基团切割下来,脱离3’端淬灭荧光基团的淬灭作用,从而发出荧光。两个探针的5’端标有不同的荧光(FAM或VIC),3’端标有MGB淬灭基团结合体。根据检测到的不同荧光,可以判断相应样本的SNP等位基因型。
Taqman分型每个位点需要2条探针,MGB探针特征如下:
(1)探针较短(~13bp);
(2)较短探针提高鉴定的准确性;
(3)小沟结合物增加了较短探针的熔点温度(Tm);
(4)TaqMan®
MGB探针对富含A/T的序列有良好的鉴定能力。


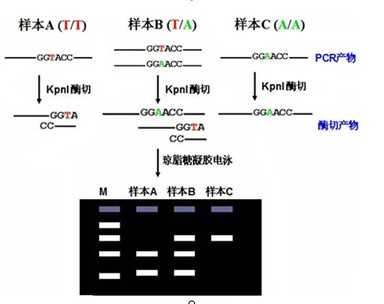
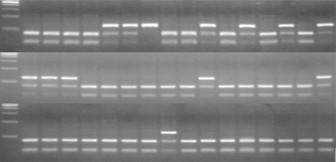
PCR-RFLP
PCR-RFLP是SNP分型最经典的研究方法,其最大优势在于,不需要有特殊的仪器,检测操作简单,费用低廉,也适合中量样本的检测;缺点是需要大量人力,耗时相对较长,不适合高通量大样本检测。
不仅能够对有酶切位点的SNP进行分型,对于不存在合适酶切位点的SNP位点,可通过在PCR引物3’端改变个别碱基引入限制性内切酶酶切位点,使该位点变成常见酶切位点,能对所有SNP位点用RFLP来分析。
SNaPshot
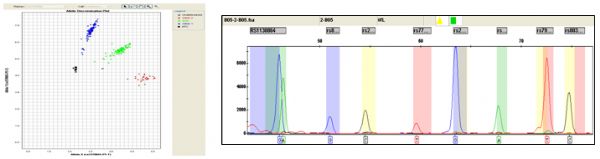
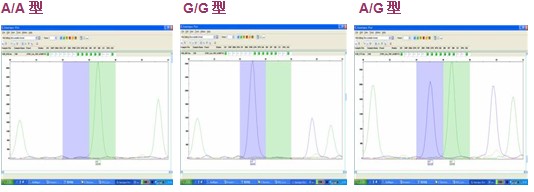
该技术由美国应用生物公司(ABI)开发,是基于荧光标记单碱基延伸原理的分型技术,也称小测序,主要针对中等通量的SNP分型项目。在一个含有测序酶、四种荧光标记ddNTP、紧临多态位点5’端的不同长度延伸引物和PCR产物模板的反应体系中,引物延伸一个碱基即终止,经ABI测序仪检测后,根据峰的移动位置确定该延伸产物对应的SNP位点,根据峰的颜色可得知掺入的碱基种类,从而确定该样本的基因型。对于PCR产物模板可通过多重PCR反应体系来获得。
SNaPshot特点如下:
(1)分型准确,该技术也称小测序技术,直接得到位点的碱基组成,检测结果直观其准确度,仅亚于直接测序。
(2)多位点同时检测,采用多重单碱基检测技术,每次反应可以检测多个SNP位点,而RFLP及TaqMan一次仅能检测一个。
(3)不受SNP位点多态特性限制,不管该位点是杂合,还是插入或缺失多态,都可以放在一个体系中检测。
(4)可以检测出受污染的样本,如果一个样本的分型峰谱偏离正常的分布,它可以提示样本可能受到污染或浓度过低,而其它分型方法则不能做到那样。

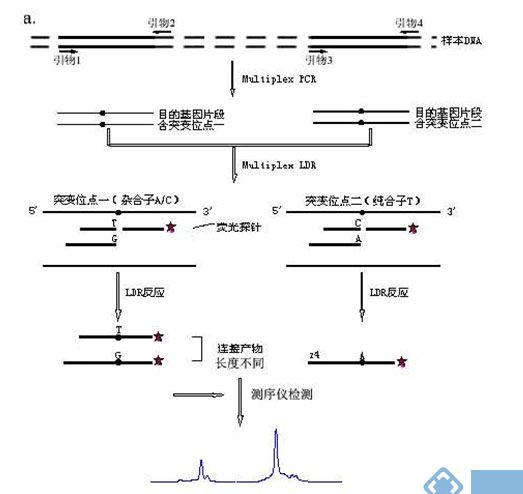
PCR-LDR
LDR是利用高温连接酶实现对基因多态性位点的识别。高温连接酶一旦检测到DNA与互补的两条寡聚核苷酸接头对应处存在着基因点突变类型的碱基错配,则连接反应就不能进行。
右边的探针与模板有一个碱基不配对,所以连接反应不能进行,没有连接产物;左边的探针与模板DNA完全互补,故进行连接反应。通过温控循环该特异性连接反应可反复进行,达到线性扩增的效果。
飞行质谱法
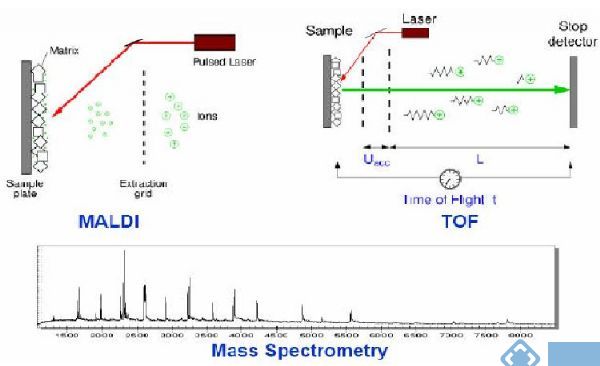
基质辅助激光解吸电离飞行时间质谱(Matrix
Assisted Laser Desorption/Ionization Time of Flight Mass Spectrometry,MALDI-TOF
MS)技术是Sequenom公司推出的世界上领先的基因分析工具,分型原理是:先通过PCR扩增目标序列,然后加入SNP序列特异延伸引物,在SNP位点上延伸1个碱基。将制备的样品分析物与芯片基质共结晶,将该晶体放入质谱仪的真空管, 而后用瞬时纳秒(10-9s)强激光激发,基质分子吸收辐射能量,导致能量蓄积并迅速产热,使基质晶体升华,核酸分子就会解吸附并转变为亚稳态离子,产生的离子多为单电荷离子,这些单电荷离子在加速电场中获得相同的动能,进而在一非电场漂移区内按照其质荷比率的不同得以分离,在真空小管中飞行到达检测器。MALDI产生的离子常用飞行时间(Time-of-Flight,TOF)检测器来检测,离子质量越小,就越快到达。利用质谱分析对质量的灵敏度特别高的特点,很容易将仅含有一个不同碱基的两段基因序列区别开,推导出SNP分型。基于MassARRAY平台的iPLEX
GOLD技术可以设计最高达40重的PCR反应和基因型检测,实验设计灵活,分型结果准确性高。根据应用需要,对数十到数百个SNP位点进行数百至数千份样本检测时,MassARRAY具有最佳的性价比,特别适合于对全基因组研究发现的结果进行验证,或者是有限数量的研究位点已经确定的情况。
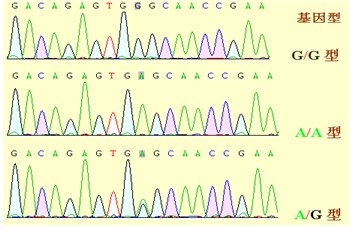
在所有SNP检测方法中,对待检测片段进行直接扩增、测序是最为准确的方法,也是SNP分析的金标准。纯合型SNP位点的测序峰为单一峰型,而杂合型SNP位点的测序峰为套峰,因而很容易将其区分开来。通过直接测序方法进行SNP检测的检出率接近100%。
通过直接测序来确定位点的基因型的分型方法准确性最高,但费用大。如果需要检测的几个SNP位点正好位于一个测序单元内(长度小于700bp),则单个位点的分型费用可显著降低,这种测序分型技术不失为一种良好选择。对于准确性要求特别高或样本量特别小(几个或几十个)的项目,这种分型技术很适合。
样本要求
■ DNA样品:浓度≥50ng/ul、体积≥20ul的总DNA样品,DNA抽提:血液20元/样本;新鲜组织30元/样本;石蜡包埋组织50元/样本
■ 血液样品:请提供≥500ul抗凝血;采用标准的采样管,抗凝剂推荐使用EDTA
■ 组织样品:请提供足量的组织样品(≥300mg)
■ 细胞(≥106 )
■ PCR产物:提供浓度≥20ng/ul、体积≥20ul(根据具体的实验位点而定)
■ SNP位点信息:SNP位点的rs号;SNP位点上下游各200bp的确切序列及SNP位点的突变类型
交付结果
1.
SNPs统计结果(Execl表格);
2. 实验过程及其涉及的电泳图、酶切图、测序峰型图;
3. 扩增和反应体系所设计的引物序列;
4. 客户所需的其他资料。