全转录组测序
产品名称: 全转录组测序
英文名称: 全转录组测序
产品编号:
产品价格: 咨询:021-64878766
产品产地: null
品牌商标: null
更新时间: null
使用范围: null
- 联系人 : 刘晴晴
- 地址 : 上海市松江区莘砖公路518号24号楼4楼
- 邮编 :
- 所在区域 : 上海
- 电话 : 137****8490 点击查看
- 传真 : 点击查看
- 邮箱 : market@cloud-seq.com.cn;liuqingqing@cloud-seq.com.cn
技术简介
真核生物转录组测序的研究对象为特定组织或细胞在某一功能状态下所能转录出来的所有RNA的总和,主要包括 mRNA、 LncRNA和环状RNA。常规的转录组分析仅通过Oligo(dT)法对mRNA进行富集和分析,会导致包括环状RNA和相当比例的LncRNA丢失,无法准确,全面的反映真实的转录组信息。云序生物转录组测序服务采用最优的样品预处理和建库方案,能够同时检测样品中的mRNA、LncRNA和环状RNA,获取最全面的转录组信息,进而针对不同类型的RNA进行表达分析,结构研究,变异研究以及全新转录本的发现。
云序优势
全面的检测所有类型的RNA:
常规的转录组测序采用Oligo d(T)法纯化带Poly(A)尾巴的RNA分子。然而很多热门的RNA分子,如环状RNA和部分LncRNA,没有Poly(A)尾巴,因此在处理过程中会被丢失掉。云序生物拥有市面上最全的,针对不同物种的去rRNA试剂盒,能够全面保留所有类型的RNA信息。
适用于降解样品:
云序生物采用特有的,可适用于降解样品的试剂盒,能够最大程度的保留降解样品中的RNA信息。尤其适合于临床样品的转录组检测。
链特异性检测:
很多基因位点会生成转录方向相反的、具有重要调控功能的反义RNA。常规的建库方法无法区分这些反义RNA,而云序生物采用链特异性的建库方法,可以准确检测反义RNA,从而提供更全面的转录组信息。
一站式服务:
客户只需提供细胞,组织,体液或总RNA,云序生物为您完成从样品准备,文库制备,上机测序到数据分析整套服务流程。
专业化的生物信息分析:
云序生物具有强大的生物信息学团队,能够满足客户的各类深入数据分析要求。
样本要求
样品类型:
组织、细胞、体液、总RNA。其它类型样品请详询。
样品量:
a) 细胞:2×107
b) 组织:100 mg
c) 体液:全血、血清、血浆2-3 ml
脑脊液:5 ml
尿液:50 ml
d) 总RNA:2 μg (OD260/280≥1.8;OD260/230≥1.5; 无明显降解,电泳检测样品特征性条带完整清晰,无明显弥散或拖尾)
样品运输及保存:
样品运输:样品置于1.5mL离心管中,封口膜封好,干冰运输。
样品保存:细胞样品或新鲜组织块可用TRIZOL或RNA保护剂处理,液氮冻存后-80℃保存;血液样品应使用EDTA抗凝,不可用肝素;RNA样品可溶于乙醇或RNA-free的超纯水中,-80℃保存,避免反复冻融。
数据分析
1、差异表达mRNA/环状RNA/LncRNA筛选
根据高通量测序结果,筛选出(Fold Change≥2,P值≤ 0.05)差异表达mRNA/环状RNA/LncRNA。
注:Fold Change代表差异程度,P值代表差异的显著性,筛选中采用的倍数变化会根据具体数据情况进行调整。
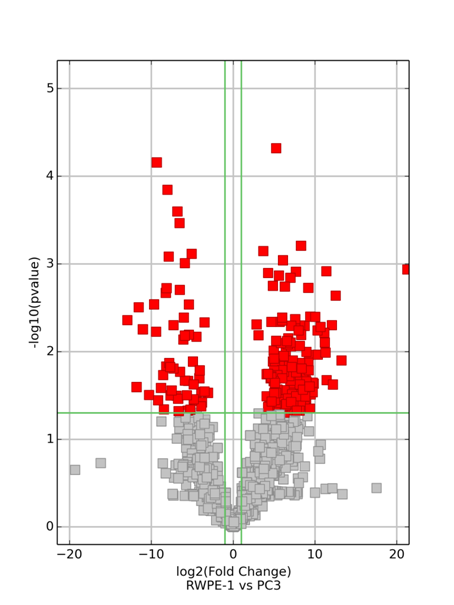
2、差异表达mRNA/环状RNA/LncRNA的聚类

3、差异表达mRNA/环状RNA/LncRNA的GO和信号通路分析
对差异mRNA/环状RNA/LncRNA的来源基因进行功能和信号通路富集分析,以便推断mRNA/环状RNA/LncRNA的功能。

4、LncRNA-mRNA共表达网络(CNC网络)构建
利用LncRNA与mRNA的共表达关系,构建LncRNA-mRNA共表达网络图,帮助发现LncRNA的靶基因,并推断LncRNA的功能。

5、竞争性内源RNA(ceRNA)分析
通过circRNA-miRNA相互作用网络分析,可以帮助解析作为miRNA海绵发挥作用的环状RNA的功能以及作用机制。

6、circRNA-miRNA-mRNA网络构建
一些LncRNA可以通过与miRNA结合,调节miRNA靶基因的表达。通过LncRNA-miRNA-mRNA关联分析,可以帮助推断作为miRNA海绵发挥作用的LncRNA以及作用机制。

案例解析
案例1:前列腺癌的全转录组分析
原文:RNA-seq analysis of prostate cancer in the Chinese population identifis recurrent gene fusions,cancer-associated long noncoding RNAs and aberrantalternative splicings
期刊:Cell Research,影响因子:15.6
本文通过转录组测序分析了14对前列腺癌组织和相应的癌旁组织,发现在不同的前列腺癌样品中在基因融合,LncRNA表达,可变剪切和点突变等方面均存在着巨大的差异。在这14个前列腺癌样品中,有3个(21.4%)存在着TMPRSS2-ERG基因融合。通过扩大样品量,还发现了2个新的基因融合CTAGE5-KHDRBS3和USP9Y-TTTY15。此外,通过表达分析发现了许多LncRNA在前列腺癌中差异表达。LncRNA和mRNA的表达相关性分析表明:LncRNA可能不仅仅在转录过程中发挥基因调控作用。

图1. 三个前列腺癌样品中的TMPRSS2-ERG 基因融合

图2. LncRNA与蛋白编码基因的表达相关性热图

图3. 前列腺癌的基因组不同位置上的体突变分布

图4. 14个前列腺癌样品中的可变剪切事件的基因组图谱
