
m5C RNA 甲基化测序服务
产品名称: m5C RNA 甲基化测序服务
英文名称: m5C RNA methylation detection
产品编号: m5C RNA 甲基化测序服务
产品价格: null
产品产地: null
品牌商标: null
更新时间: 2024-06-27T08:51:38
使用范围: null
- 联系人 : 高翔
- 地址 : 上海市松江区新桥镇莘砖公路518号28幢402室
- 邮编 : 201612
- 所在区域 : 上海
- 电话 : 186****5001 点击查看
- 传真 : 点击查看
- 邮箱 : sales@neo-bio.cn
一、m5C RNA 甲基化测序
m5C RNA 甲基化测序是一种用于研究 RNA 甲基化状态的生物技术,其中 m5C 代表 5-甲基胞嘧啶,这是一种在 RNA 分子上发现的修饰。用重亚硫酸氢盐(Bisulfite)处理核酸是一种常用的方法,用于将未甲基化的胞嘧啶转化为尿嘧啶,而甲基化的胞嘧啶保持不变,从而可以通过测序区分甲基化和非甲基化的胞嘧啶位点。
二、服务目标
1. 确定 RNA 分子上的 5-甲基胞嘧啶(m5C)位点
2. 研究 RNA 甲基化在基因表达调控中的作用
3. 揭示不同状态的样本中 RNA 在C碱基上的甲基化变化
三、实验原理
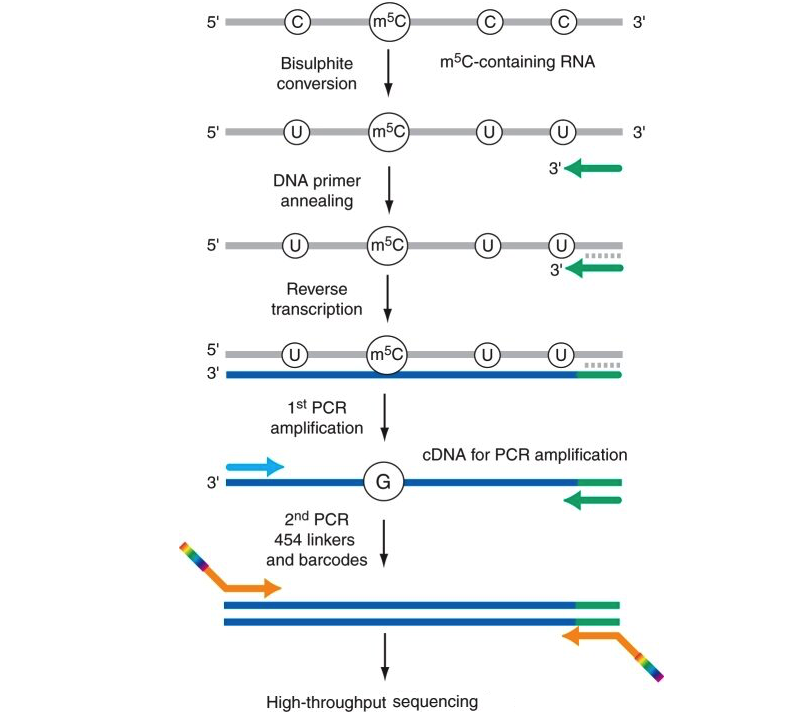
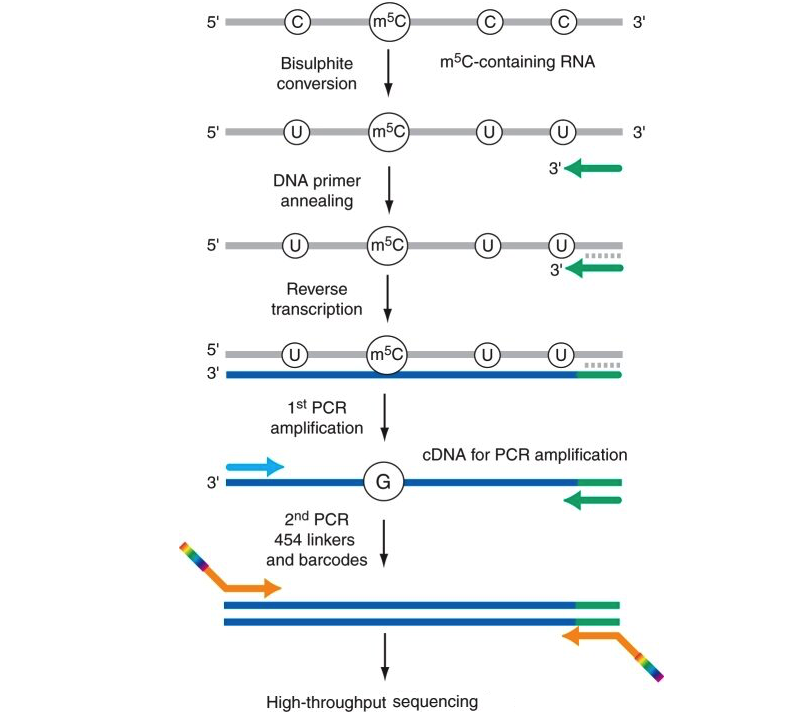
图一、Bisulfite处理RNA,转化C碱基为U碱基的原理示意图
重亚硫酸氢盐处理在RNA甲基化测序中的原理是基于重亚硫酸氢盐的化学性质,它可以将RNA中的未甲基化的胞嘧啶(Cytosine, C)转化为尿嘧啶(Uracil, U),而甲基化的胞嘧啶(5-methylcytosine, 5mC)则不会被转化。再经过逆转录和PCR扩增,U碱基会被转化成T碱基。在测序完成之后,生物信息分析中根据原来是C的碱基是变转变为T碱基,来判断原来RNA中的C碱基是否发生了甲基化。
四、服务流程
- 样本收集与处理
- 客户提供 RNA 样本
- 确保 RNA 样本质量与纯度
- RNA 质量评估
- 使用凝胶电泳或生物分析仪评估 RNA 完整性和纯度
- 重亚硫酸氢盐处理
- 使用重亚硫酸氢盐处理 RNA,将未甲基化的胞嘧啶转化为尿嘧啶
- 逆转录
- 使用特定的逆转录酶合成 cDNA
- 文库构建
- 利用合成的 cDNA 构建测序文库
- 高通量测序
- 使用高通量测序平台进行测序
- 数据分析
- 利用生物信息学工具进行数据质量控制
- 识别 m5C 位点
- 进行甲基化水平的定量分析
- 结果解释与报告
- 提供详细的分析报告,包括甲基化位点列表、甲基化水平
- 更进一步的个性化分析,可向逆耳公司具体咨询
五、客户样本准备
- 组织
l 样本的采集:
1) 应使用新鲜组织,体积要小,尽量保证长宽高不超过0.5cm,以绿豆大小为参考。
2) 去除不需要的组织类型,如结缔组织、脂肪组织等。
l 清洗和干燥:
1) 迅速用预冷的RNase-free水配制的1×PBS或生理盐水清洗组织表面,去除血迹和污渍,然后用无尘纸巾吸干。
l 冷冻:
1) 处理好的组织应迅速放入预冷的RNase-Free的冻存管中,并在液氮中速冻1小时后转移到-80°C长期保存,避免反复冻融。
2) 在冻存管上清楚地标记样本的名称、收集的日期等信息。
l 保存:
1) 如果实验室条件有限,可以使用RNAlater等组织保存液,按照操作说明进行,使样本完全浸没在液体中,4°C保存过夜,然后转移到-80°C保存。
- 培养细胞
l 细胞培养:
1) 确保细胞在适宜的条件下生长,显微镜下观察细胞以确定其生长状态良好。
l 收集细胞:
1) 对于贴壁细胞,弃去培养基,小心加入与培养基等体积的无酶水配制的1×PBS洗涤细胞,然后弃去PBS,重复一次。
2) 对于悬浮细胞,选取生长状态良好的细胞悬液,离心以收集细胞沉淀。
l 裂解细胞:
1) 加入适量的裂解液(如Trizol)反复吹打至充分裂解。裂解充分的标准是吹打时液体不粘稠,流动性好。
l 收集裂解物:
1) 将裂解后的细胞转移到耐低温的螺纹口冻存管中。
l 冷冻保存:
1) 将含有细胞裂解物的冻存管迅速置于液氮中冷冻10分钟左右,然后转移到-80°C长期保存。
2) 在冻存管上清楚地标记样本的名称、细胞类型、收集日期等信息。
l 避免反复冻融:
1) 在RNA提取前避免样本的反复冻融,因为这会破坏RNA分子。
- RNA样本
l 将纯化好的RNA样本储存在-80°C或更低的温度下,避免反复冻融。
l 在冻存管上清楚地标记样本的名称、细胞类型、收集日期等信息。
- 样本寄送:
l 干冰进行样品寄送到逆耳生物公司实验室,干冰消耗量为5kg/天,干冰采用厚实的泡沫箱进行打包。
参考文献:
1、 Matthias Schaefer, Chapter Fourteen - RNA 5-Methylcytosine Analysis by Bisulfite Sequencing [J] Methods in Enzymology, Volume 560, 2015, Pages 297-329
2、 Matthias Schaefer, Tim Pollex, et al. RNA cytosine methylation analysis by bisulfite sequencing [J] Nucleic Acids Research, Volume 37, Issue 2, 1 February 2009, Page e12
3、 Yuri Motorin, Frank Lyko, et al. 5-methylcytosine in RNA: detection, enzymatic formation and biological functions [J] Nucleic Acids Research, Volume 38, Issue 5, 1 March 2010, Pages 1415–1430
