根据客户需求,提供个性化服务,是微基生物的一大特色!由于研究需求,利用通用的16S rDNA /18S rDNA/ ITS进行高通量测序分析(NGS)并不能满足广大客户对某一类特定微生物的详细分类需求,越来越多的客户开始着力于对某些特定功能的靶基因进行建库研究。
采用16S rDNA /18S rDNA/ ITS通用基因进行微生物多样性分析,所研究的是样本中的全部微生物,并不能够对特定功能的某些种属内微生物进行详尽的分类研究。通过对某些特定功能的靶基因高通量测序分析,能够对所某些种属内的目的微生物进行详细的分类研究,更加清晰的分析目的微生物的物种信息及生态分布情况。
传统的靶基因建库方法,直接采用PCR扩增产物构建靶基因文库,再进行Sanger测序,整个研究过程耗费较长时间及人力,测序费用昂贵;利用高通量测序对靶基因建库分析,数据量大幅度提高,费用却大幅下降,且能对样本中靶基因的分布进行定量分析。
客户可根据自己的需求,提供所需研究的靶基因的特异引物,我们来负责构建适合illumina miseq平台或是Roche 454平台测序所需的文库。在数据分析时,通常情况下,参照数据库(reference database)都是通用库,比如silva的16S/18S数据库。但是对于靶基因,在没有通用库的情况下,一般上千条序列即可构建靶基因的参照数据库,我们可根据客户提供的序列信息进行自建库的构建,为序列的后续数据生物信息学分析提供资料。
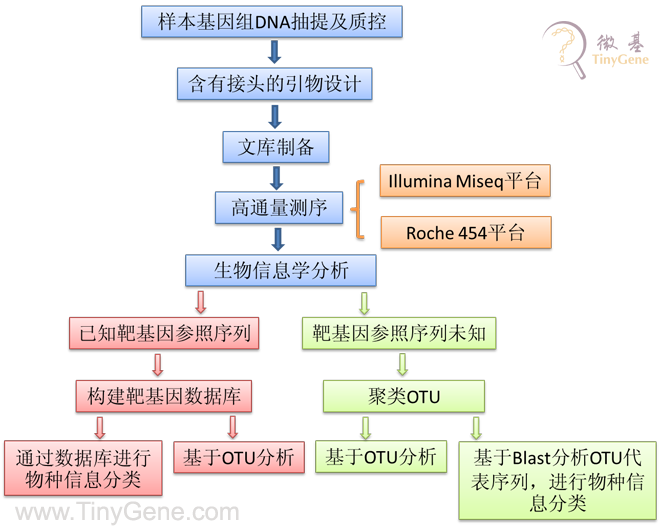
技术路线:
1、定鞭金藻:
定鞭藻门是海洋原生生物的一个关键门。近年来,更多研究者着力于定边藻门微生物的研究,他们利用核糖体DNA大亚基(LSU rDNA)的一对特异引物,基于Roche 454测序平台对靶基因进行建库分析。
研究案例:
Lucie Bittner等基于核糖体DNA大亚基(LSU DNA)D1和D2区域的定鞭金藻特异引物和Roche 454测序平台对Naples Bay中未培养的定鞭金藻多样性分布模式进行分析。本文通过自己构建数据库(包括1290个环境定鞭金藻的环境克隆文库和172个培养定鞭金藻菌株的序列),将测序结果与之进行比对分析。该文章也阐述了在研究大量未知单细胞真核微生物多样性中在技术和理论上的固有障碍。
参考文献:
Diversity patterns of uncultured Haptophytes unraveled by pyrosequencing in Naples Bay
所得主要结果:
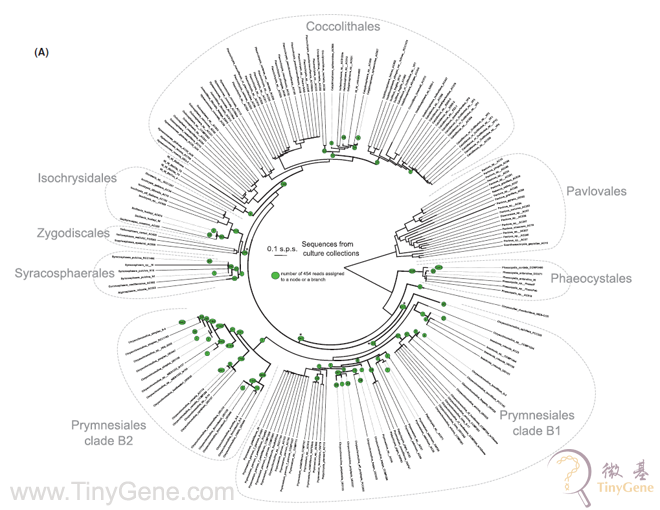
(A)将13501个reads与172个培养定鞭金藻序列一起构建系统发育树图谱,节点或是分支处的绿色圆点代表reads的数目。该树形图谱形象的展示所得结果中可以分析出定鞭金藻的种类。
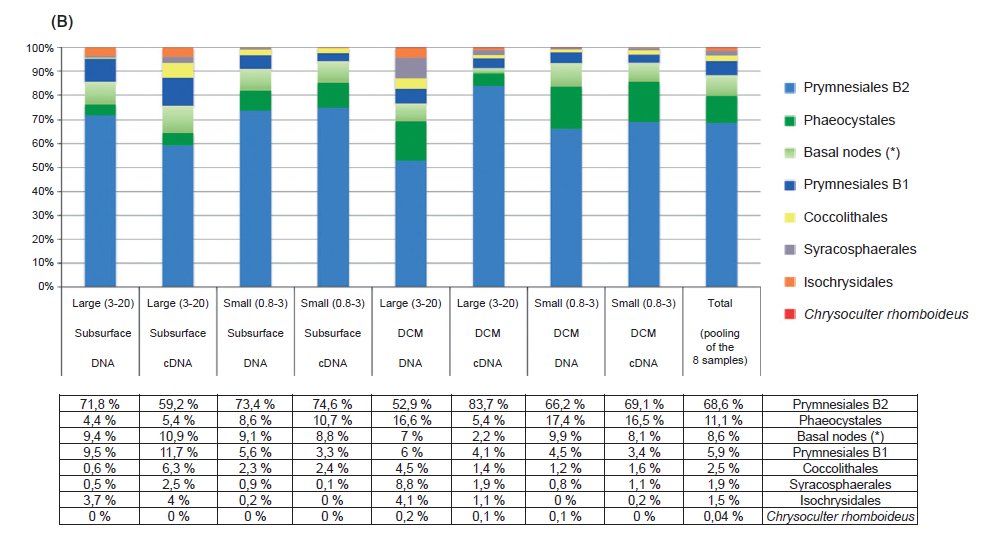
(B)定鞭金藻分类后,每个样本中各物种所占的百分比。清楚的了解到Naples Bay定鞭金藻的物种分布情况。
2、硝化菌:
硝化细菌在促进水域生态系统的氮循环、保持健康水产养殖环境方面发挥着巨大作用。硝化作用(Nit rification) 是将氨氧化成亚硝酸(盐) ,再氧化成硝酸(盐) 的过程。前者主要是由自养性的亚硝酸菌或氨氧化菌(Ammonia oxidizing bacteria,AOB) 完成,后者是由硝酸菌或亚硝酸盐氧化菌(Nit rite oxidizing bacteria,NOB) 完成。氨氧化菌还包括氨氧化古菌(Ammonia oxidizing archaea,AOA)
除了16S rRNA 基因以外,催化氨氧化的功能基因氨单加氧酶α亚基基因( amoA ) 也被广泛用做自然环境中AOB、AOA 遗传多样性及丰度研究的分子标记。氮氧化中的关键酶(catalytic subunit of nitrite oxidoreductase ,NXR)的编码基因nxrA被用作研究NOB多样性。
研究案例:
Hang-Wei Hu等利用454测序技术,通过古菌的16S rRNA gene和 AOA 、AOB的功能基因amoA研究来自不同区域的65份土壤样本中氨氧化微生物的相对丰度、多样性及群落组成情况,研究结果表明,氨氧化微生物的分布与土壤的pH值有密切相关性。
所得主要结果:
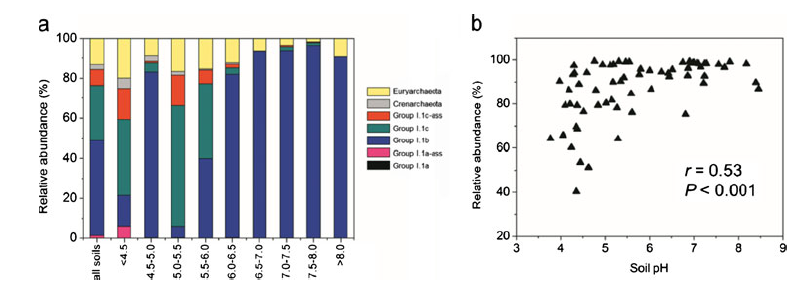
对不同pH值条件下氨氧化微生物中各物种分布相对丰度进行统计,统计结果如图,氨氧化微生物的群落组成与土壤的pH值有密切的关系。
3、反硝化菌
随着现代研究的不断发展,更多的反硝化微生物从土壤、沉积物、水环境中分离得到。由于反硝化微生物是一大类生物类群,现已被发现广泛存在于细菌、真菌及古菌中,因此传统经典的16S rDNA方法就不适合反硝化菌的生态学研究。因此,一些功能基因,如反硝化酶编码基因常被选作分子探针和引物设计的模板用于多种生态环境下微生物体系与种群结构的分析。如:硝酸盐还原酶编码基因Nar、亚硝酸盐还原酶编码基因Nir、氧化还原酶编码基因Nor、氧化亚氮还原酶编码基因Nos。
研究案例:
法国研究者Philippot 等利用高通量测序技术分析反硝化微生物,采用了 454 测序平台对反硝化菌的nosZ 基因进行了测序分析,发现了微生物多样性的降低会显著影响反硝化微生物多样性以及群落结构。
参考文献:
Loss in microbial diversity affects nitrogen cycling in soil. ISME J. 2013
所得主要结果
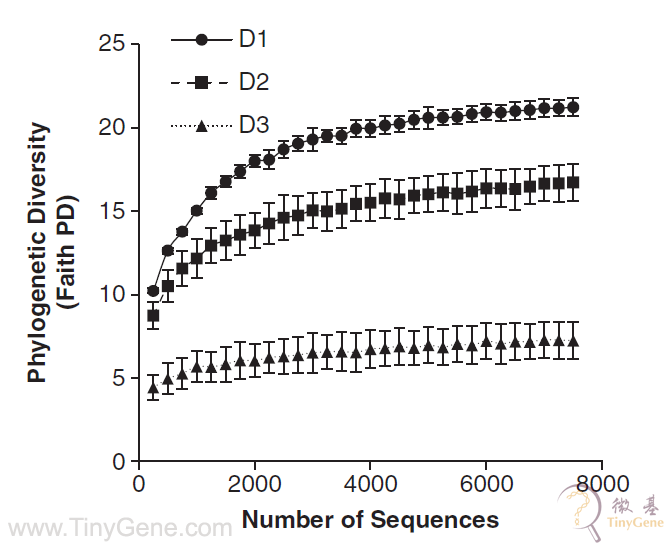
(1)将样本稀释处理,D1为原浓度、D2为稀释103倍、D3为稀释105倍,进行Faith’s PD稀释曲线绘制,发现反硝化微生物的物种多样性随稀释倍数升高而降低。
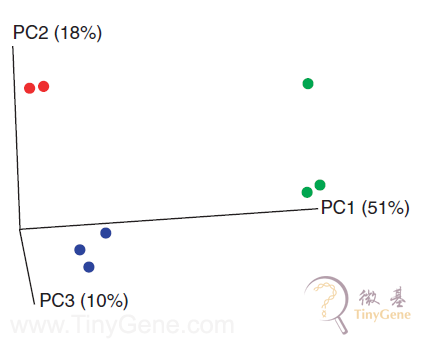
(2)通过unweighted UniFrac distances进行PCoA分析,发现不同稀释条件下,微生物群落分布存在显著地不同。
研究案例
芬兰学者Palmer K利用了 454 高通量测序技术,研究了受冻裂影响的北极苔原泥炭土反硝化微生物特征与 N2O 排放的相互关系,对 功能基因narG、nirK/nirS 以及 nosZ 进行了测序分析和定量分析,结果发现高 N2O 排放量都和土壤中的一些特殊反硝化微生物有关,说明N2O 排放量的大小和土壤中反硝化组成是有对应关系的。
4 硫酸盐还原菌
硫酸盐还原菌 (Sulfate-Reducing Bacteria,简称SRB) 是一种厌氧的微生物。广泛存在于土壤、海水、河水、地下管道以及油气井等缺氧环境中。研究表明在无氧或极少氧情况下,它能利用金属表面的有机物作为碳源,并利用细菌生物膜内产生的氢,将硫酸盐还原成硫化氢,从氧化还原反应中获得生存的能量。据不完全统计,SRB目前已有12个属40多个种 ,SRB的分类学研究进展比较缓慢。生理学上分为两类:将硫酸盐还原为硫化氢,将硫酸盐还原为硫。
SRB 硫酸盐还原途径中的腺苷酰硫酸还原酶( adenosine-5′-phosphosulfate reductase,Apr) 基因和异化型亚硫酸盐还原酶( dissimilatory sulfite reductase,Dsr) 基因是它常用的遗传标记物。
研究案例
wenjing jia等采用半巢式PCR技术,基于Roche 454平台对CD、IBS、UC患者与健康人粪便中硫酸盐还原菌的多样性及分布进行研究。发现两个主要的物B. wadsworthia和Desulfovibrio piger在健康人群占全部SRB的93.5%,但是在所有患者中的比例都是相对较低的。
所得主要结果
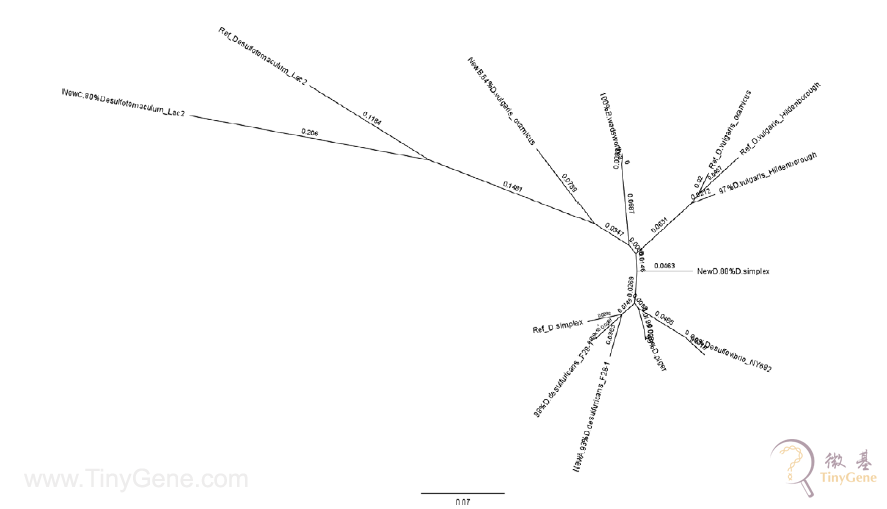
(1)在7个分组中,4中SRB所占的百分比的统计图如下,SRB中两个主要物种在健康人中所占比例较6个患者组相对较高。

(2)本研究发现一致序列共包括9个种系型,与8 个参照物种一同构建进化树。形象显示分析结果与参照物种之前的关系。 Bilophila wadsworthia, Desulfovibrio piger, D. desulfuricans F28-1 and Desulfovibrio NY682它们与参照物种的标签在图上重合,以至于很难区分它们。