动植物基因组de novo测序
产品名称: 动植物基因组de novo测序
英文名称: Plant & animal genome de novo sequencing
产品编号:
产品价格: 询价
产品产地: null
品牌商标: null
更新时间: null
使用范围: null
- 联系人 :
- 地址 : 上海市浦东新区国际医学园区康新公路3399弄3号楼
- 邮编 : 201203
- 所在区域 : 上海
- 电话 : 159****9102 点击查看
- 传真 : 点击查看
- 邮箱 : marketing@majorbio.com
产品介绍
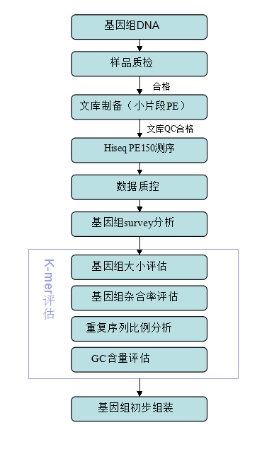
基因组survey
在正式启动基因组项目之前,基于小片段文库的低深度测序数据,通过K-mer分析,可以有效地评估基因组大小、GC含量、杂合度高低以及重复序列含量等信息,是全面了解某一物种基因组特征的有效方法,为后续全基因组de novo测序及组装策略的制定提供依据;此外,通过基因组survey分析也可对基因组进行初步组装,开发SSR标记等分析。
技术路线
技术参数
|
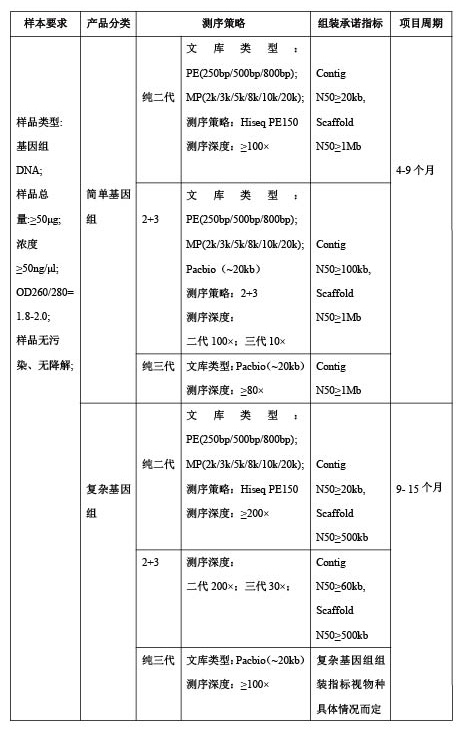
样本要求
|
策略 |
服务周期 |
|
样品类型:基因组DNA; 样品量:浓度≥30ng/μl,DNA总量≥4μg;OD260/280介于1.8-2.0之间并确保DNA无降解、无污染,切忌反复冻融; |
测序平台:Hiseq2500/4000 文库:250bp/500bp 数据量:≥40× |
40天 |
基因组de novo测序
基因组de novo测序也叫做基因组从头测序,是指不依赖于任何已知基因组序列信息对某个物种基因组进行测序,然后应用生物信息学手段对测序序列进行拼接,最终获得该物种基因组序列图谱。从基因组水平上对物种的生长、发育、进化、起源等重大问题进行研究,加深我们对测序物种的认识,在新基因的发现、物种改良等方面发挥巨大作用。
根据基因组复杂程度,可以分为简单基因组和复杂基因组:
简单基因组:主要指重复序列比例低于50%的单倍体或高纯合二倍体(杂合率低于0.5%),如大部分哺乳动物、鸟类以及栽培作物等。
复杂基因组:主要指重复序列比例高于50%,杂合率大于0.5%的二倍体或多倍体,如大部分林木、水产类以及昆虫等。
美吉优势
拥有标准化实验室和高通量测序技术平台,实验周期短,质量可靠;
拥有Illumina HiSeq 2500/4000、MiSeq、Pacbio等多种高通量测序平台;
技术人员经验丰富,可根据客户需求提供实验方案、解决实验问题、分析实验结果;
拥有专业的生物信息分析团队和大型计算机,可提供个性化的生信分析服务;
提供合作论文撰写及发表;
技术路线

生信分析
生信分析流程:
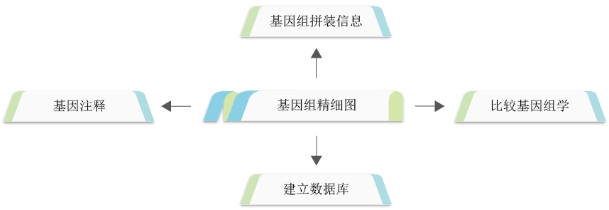
生信分析内容:
基因组拼装统计
提供基因组拼装的基本信息,包括原始数据统计、测序覆盖度统计、Contig N50大小、Scaffold N50大小、基因组大小、GC含量等组装结果统计。
基因结构注释
包括基因预测、重复序列分析及non-coding RNA预测等。
基因功能注释
Nr注释;Interpro注释;GO和KEGG注释;SwissProt及TrEMBL注释
比较基因组学及进化分析
基因家族鉴定 & 基因家族收缩与扩张;物种进化树构建 & 物种分歧时间估算;基因组共线性分析 & 全基因组复制事件分析;正选择基因的鉴定及功能富集分析;
建立数据库
建立符合国际标准且具有良好兼容性的基因组数据库,实现基因组数据的查询与共享。
技术参数
送样要求:
1. 样品类型:动植物组织或者DNA
2. 样品DNA需求量:
250-500bp小片段PE文库> 2µg;
3-5K大片段MP文库> 10µg;
5-10K大片段MP文库>40µg;
10-20K大片段MP文库>60µg;
Pacbio文库>10µg;
3. 样品浓度:
小片段文库>50ng/µL;
大片段文库>100 ng/µL。
4. 样品纯度:OD260/280=1.8-2.0并确保DNA无降解,无污染。
5. 样品保存期间切忌反复冻融,送样时请使用冰袋或干冰运输。
案例解读
案例一:草鱼基因组测序揭示其进化与草食适应性[1]
草鱼是我国重要的淡水养殖鱼类,也是世界范围内最重要的淡水养殖品种之一,其产量约占全球淡水养殖总量的16%。基因组测序组装得到0.9Gb雌性草鱼基因组和1.07Gb雄性草鱼基因组序列。分析显示草鱼与斑马鱼的分化时间发生在4900-5400万年前,与斑马鱼相比草鱼发生染色体融合现象。转录组分析发现肝脏中甲羟戊酸通路和类固醇生物合成通路被激活与草鱼食性转变有关。
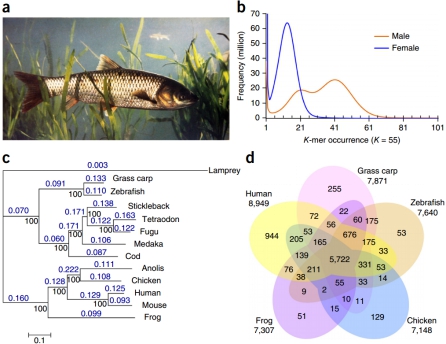
图1 草鱼基因组组装与进化
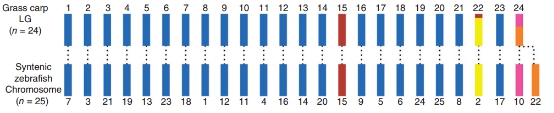
图2 草鱼与斑马鱼核型图
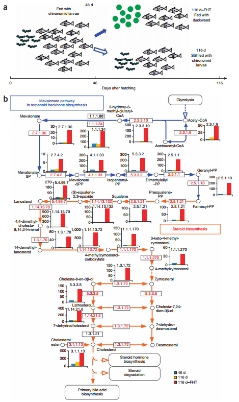
图 3 转录组分析揭示草鱼食性转变
案例二:菠萝基因组测序和CAM光合途径进化研究[2]
人类培育菠萝的历史已经超过了6000年,开始于现在的巴西西南部和巴拉圭东北部。全球80多个国家每年生产约2500万吨的菠萝水果,总产值接近90亿美元。基因组测序组装得到382Mb,占基因组72.6%;共27,024个基因,其中89%是完整的。染色体核型进化分析显示菠萝基因组经历了σ和τ两次全基因组复制事件(图1)。CAM途径分析显示菠萝景天酸代谢光合作用不是通过全基因组复制或串联基因复制产生的重复基因演化而来(图2)。

图1 菠萝基因组两次全基因组复制事件
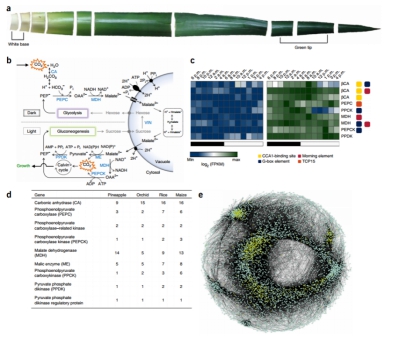
图2 菠萝景天酸途径的演化
参考文献
[1]Wang Y, Lu Y, Zhang Y, et al. The draft genome of the grass carp (Ctenopharyngodon idellus) provides insights into its evolution and vegetarian adaptation. Nature Genetics, 2015, 47(6): 625-631.
[2]Ming R, VanBuren R, Wai C M, et al. The pineapple genome and the evolution of CAM photosynthesis. Nature Genetics, 2015, 47(12): 1435-1442.
常见问题
(1)动植物基因组de novo测序之前为什么必须先进行survey?
动植物基因组大小(几十兆-几百G)、结构复杂度(重复序列、杂合率),染色体倍性等差别较大,需要根据基因组情况制定合适的测序策略,如果直接测序组装,组装质量可能会非常差。
(2)植物基因组测序对样品取样有什么特殊要求?
最好选择黑暗无菌条件下培养的黄化苗或组培样品取样;建议采用纯合二倍体或单倍体材料。
(3)动物基因组测序对样品取样有什么特殊要求?
样品提取应选用肌肉、血液等脂肪含量较少的部位,并尽量选用同一个体进行取样。如果物种体积较小,单个个体所提取的 DNA 量不足一次测序反应,在保证量的情况下,应当尽量减少物种的个数,以减少个体差异性对后续拼接的影响。样品建议采用单倍体或纯合二倍体材料。
(4)如何检测最终组装结果的准确性?, ,
目前,主要可以通过以下几种方法来检验基因组组装的准确性。
①通过构建BAC或Fosmid文库,并进行常规测序,将所得序列与拼接好的Contigs做比对以判断基因组组装的准确率。同时也可以与CEGMA数据库中核心基因进行比较查看测序物种预测得到的基因完整性评估基因组的准确性。
②将已知的基因序列与拼接好的Scaffolds做比对,查看两者是否吻合,吻合度越高,表明基因组组装越好。而且已知的基因序列越多,评价结果越可靠。
③估计组装后基因组的单碱基准确度,如果95%以上的基因组单碱基覆盖度超过20×,则认为该基因组的单碱基准确度较高。