三代微生物多样性是基于PacBio SequelⅡ测序平台,利用单分子实时测序的方法,对原核生物16S的全部V1-V9可变区域或真核生物的18S高变区域或ITS区域进行全长扩增,不仅能提高物种鉴定的分辨率,还能提高样本中微生物组成鉴定的精确度,从而更全面的反应微生物的群落结构。
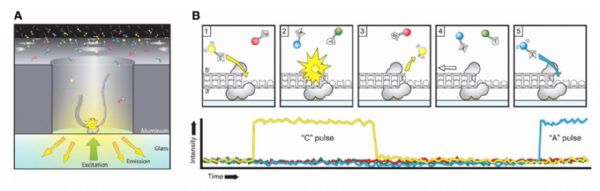
利用与二代测序相同的边合成边测序的原理,以SMRT芯片为载体;模板与特殊聚合酶结合后,4种不同荧光标记的脱氧核糖核苷三磷酸(dNTP)通过布朗运动随机进入检测区与聚合酶结合,按DNA模板序列延伸;通过检测单个DNA分子的合成所产生的信号来进行测序,也可通过检测相邻两个碱基的测序时间,来检测一些碱基修饰情况。
图1. PacBio测序原理[1]
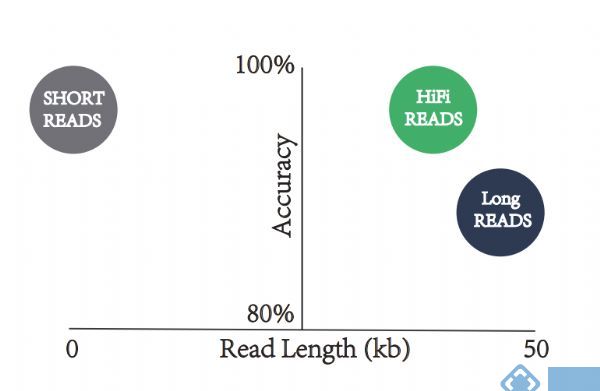
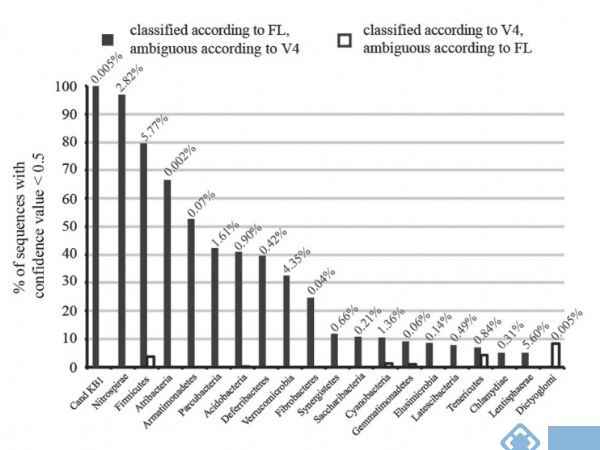
于二代测序只能获取1~2个高变区短读长序列而言,三代测序能一次性测到所有高变区,同时也可以避免不同引物带来的偏好性,从而提高微生物群落的分类学分辨率。
图2. 测序长度和准确度
图3. 物种鉴定结果
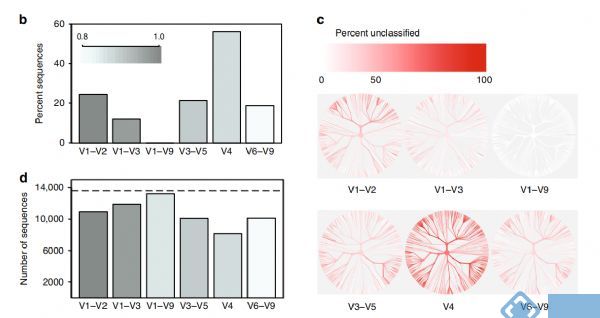
图4. 微生物群落分布
图5. 技术流程图
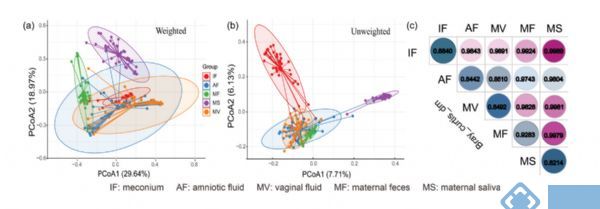
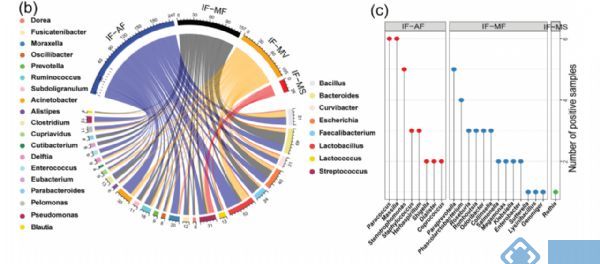
研究小结:早期微生物与成年后疾病的发生发展有一定的关系,研究发现子宫、胎便、羊水和胎盘都有微生物存在,那么婴儿胎便微生物来源与母体不同来源的样本的关系如何呢?本文以39对母婴的产妇样本(羊水、粪便、阴道分泌物和唾液)和婴儿胎便样品为研究对象,基于PacBio三代全长测序技术进行16S rRNA检测。检测发现不同类型样本间α多样性和β多样性存在特异性;同时除发现不同类型样本各自的一些优势菌(其中羊水和阴道分泌物的优势菌为Lactobacillus和Curvibacter,胎便的优势菌为Bacillus 和Escherichia/Shigella,产妇粪便的优势菌为Bacteroides和Faecalibacterium,产妇唾液的优势菌为Streptococcus和Prevotella)外,还发现了他们间共有OTUs,其中胎便菌群与羊水菌群的共有OTUs多于产妇粪便菌群和阴道菌群,由此推断羊水菌群相比其他位点贡献更大。此外,发现剖腹产和顺产胎儿的胎便和羊水微生物群落结构无显著差异。
图6. 文章部分结果
参考文献:
文献下载链接:
https://pan.baidu.com/s/1eNadtYArpOiFy3SUPpPrjQ
提取码:YG45