精准医疗,也被称为个性化医学,是指使用病人特定的分子特征来指导疾病诊断、监测病情发展与治疗应答、或用于确定药物开发新靶点的医疗模式。
精准医疗原则的应用使癌症治疗领域取得了长足的进步,并有望显著改变多种病症的治疗,包括代谢和心血管疾病、自身免疫和炎症性疾病,以及神经发育和神经退行性疾病。在这篇文章里,我们将回顾精准医疗的基本理念和面临的挑战,并讨论表观基因组和染色质生物标志物如何成为推进这一重要研究的关键。
精准医疗的必要性
就历史观点而言,大多数治疗方法都是面向广大患者群体使用的。然而,疾病的病因十分复杂,通常由遗传、环境和生活方式等相互作用引起。因此,治疗结果可能差别很大,患者可能完全消除或减轻症状,也有可能面临着中度或严重的失败治疗,产生更多副作用或病情进展没有变化,而精准医疗是解决这一问题的潜在方案1,2。精准医疗的首要目标是利用具体病患的分子特征来了解如下多个方面的信息:
● 诊断信息。该病的特征是什么?
● 患者分层。我们能否利用生物标志物来完善复杂疾病的亚型?
● 疾病预后。该病有多严重?它的病程有多快?
● 治疗选择。对于特定的病患,最佳的治疗方案是什么?
● 治疗应答指标。药物的效果是否符合预期,还是应该改变剂量,尝试另一种治疗方法,或者用另一种药物作为补充?
● 新药开发。我们能否确定新的治疗靶点?
在寻找这些问题答案的过程中,相当一部分的研究经费被用于确定合适的治疗靶点和生物标志物。下面,我们将着重介绍当前方法、临床上的成功案例以及精准医疗研究的发展前景。
精准医疗的基本原则
● 基于遗传变异的方法
尽管精准医疗的概念早在20世纪60年代就已经存在,但直到21世纪初人类基因组计划完成后,这一运动才获得了真正的推动力1。这一壮举使得科学家们能够对人类遗传变异(如突变和单核苷酸多态性)进行大规模研究,并确定其与独特表型或疾病的关联性3。这些项目被统称为全基因组关联分析(GWAS),确定了数十万种疾病和性状的风险变异4。
在某些情况下,识别疾病相关风险变异揭示了潜在的疾病生物学机理,这反过来又能够推动医疗发展。除此之外,GWAS确定了可选择的适合患者治疗方法的生物标志物。下面我们重点介绍几个关键应用:
案例研究1:克罗恩病和IL-12/IL-23抑制剂
在克罗恩病(Crohn’s disease)中,GWAS发现细胞因子IL-12和IL-23与疾病发展相关联5。针对这些分子促炎作用的基础研究促进了靶向这一途径的免疫治疗药物实验的成功6。
案例研究2:可用于治疗选择的肺癌生物标志物
DNA测序已经发现了多种肺癌相关的基因变异7,其中7种已被FDA批准用于治疗8。这使得医生可以利用基因检测来选择治疗方法,并希望借此改善患者的预后。
然而,仅识别疾病相关的风险变异可能会导致不完备的病因学观点。因此,为了充分利用这些发现推进精准医疗的发展,必须了解其功能性结果。例如,基因组中蛋白质编码区的变异可能影响蛋白质的结构、功能和表达。如囊性纤维化疾病中每一种变异都代表着一种不同的分子结果,可能需要以不同的方式对待。
案例研究3:遗传风险变异为囊性纤维化的治疗提供信息
多种遗传变异与囊性纤维化有关。根据致病变异的不同,两种对CFTR蛋白具有不同作用机制的药物(帮助运输到细胞表面或离子通道功能)可单独或联合使用9-11。
值得注意的是,90%以上经GWAS确认的变异都位于基因组的非编码区12,而非编码区富含细胞类型特异的、能够激活基因表达的顺式调控元件(即增强子)13。因此,许多研究人员专注于研究疾病风险变体如何影响转录调节,而不是蛋白质结构或功能14-16。
目前面临的挑战在于了解靶标基因是什么。非编码区可以通过直接和间接的机制跨越基因组距离发挥作用;因此,与疾病变异最接近的基因不一定是因果决定因素17,18。
● 基于基因表达的方法
研究人员发现,在不同的疾病状态下,包括癌症、自身免疫性疾病、神经退行性疾病、心血管疾病和其他疾病等,基因表达都发生了很大的变化19-23,这突出了通过全基因组调查方法发掘疾病的转录特征的必要性。基因表达研究,包括微阵列和RNA测序方法在内,已经帮助研究者确定了许多疾病的亚型。
案例研究4:乳腺癌患者分层和个性化治疗选择
归功于基于精准医疗的诊断和治疗的应用,乳腺癌患者的五年生存率已大幅提高24(从1975年的76%提高到现在的90%以上25)。乳腺癌可以通过多种信号受体的表达进行分类,为诊断、选择合适的治疗组合和复发风险提供信息。例如,酪氨酸激酶HER2(HER2+)表达阳性的患者通常使用曲妥珠单抗(trastuzumab),一种靶向HER226的单克隆抗体。将HER2状态等分子分类与细胞分化和增殖的定量测量相结合,也为患者的预后提供了信息27。这些只是大量研究结果中的其中几个例子,它们推动了该领域的发展,帮助医生治疗并给予了乳腺癌患者及其家庭支持。
基于基因表达的个性化医疗研究仍在进行,既要进一步完善有既定生物标志物的癌症亚类,如乳腺癌28,也要完善没有生物标志物的癌症亚类,如一些脑癌29。这些发现将产生新的生物标志物和治疗靶点,可能拯救许多生命。
当前精准医疗方法的缺陷
在上文中我们重点介绍了精准医疗领域在多方面的临床成功案例。尽管精准医疗已经取得了这些显著的成就,但GWAS和转录组学往往无法产生与临床相关的生物标志物或药物靶点,令许多疾病难以诊断和治疗。这可以归因于许多悬而未决的问题:
● 哪种变异是疾病的真实原因?
这些变异很少单独发生。事实上,变异通常是在被称为单倍型的大块DNA中成组遗传的,这使得我们很难知道哪些变异(如果确实存在的话)可能会影响疾病状态。
● 遗传和环境的影响如何交叉作用?
事实上,一些遗传变异的外显率受到生活方式30或环境因素31的调节,如服用某些处方药、运动和环境污染,其基因调控影响和疾病风险与暴露程度相关。
● 环境因素是否更可能是主要原因?
一些与疾病相关的基因调控变化与遗传变异完全无关。相反,这些基因调节变化的根本原因——与从过敏32到低出生体重33到心脏病34等状态相关——取决于环境暴露。
● 基因表达是否涵盖了我们需要知道的一切?
目前研究异常RNA表达的方法提供了细胞内当前情况的快照。它们不能提供有关细胞“准备做什么”的信息,如治疗引起的遗传程序的变化或抗药性的发展。这种机制信息的缺乏留下了许多关于治疗应答和复发可能性的开放性问题,而这些问题对个性化护理具有极大的参考价值。
● 为什么我们只关注蛋白质编码区?
基因表达和遗传变异是疾病的主要指标——但它们只是影响因素的一部分。事实上,人类基因组中只有不到1%编码蛋白质35。基因调控主要由非编码区控制,非编码区可以作为增强子,形成染色质环,结合转录因子,或招募表观遗传修饰酶和阅读器蛋白。此外,许多疾病风险变异也富集在非编码DNA中。因此,非编码DNA和基于染色质的机制代表了大量未开发的生物标志物和药物靶点。
遗传、环境、转录和染色质的融合使得疾病表型发展十分复杂(Figure 1)。目前的方法通常无法考虑这些不同的因素,使得人们难以预测疾病进展并对治疗作出及时的反应。
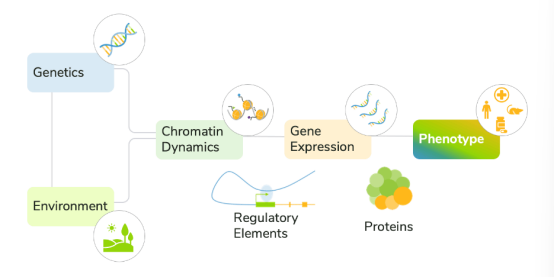
Figure 1 Environmental and molecular factors together drive disease development.
将表观基因组学纳入精准医疗
表观基因组可能是确定新型生物标志物和药物靶点以推进精准医疗的关键。表观基因组包括复杂的分子代码,能够调节染色质结构和功能以控制基因表达。因此,对表观基因组进行的研究可以作为一个重要的分子工具,帮助我们了解由遗传变异、环境和治疗导致的基因表达变化。
为此,染色质修饰,包括DNA甲基化和组蛋白翻译后修饰(PTMs),已经为基因调控程序提供了显著的见解,并正在成为精准医疗研究的重要组成部分。
● DNA甲基化
从历史观点上说,绘制DNA甲基化图谱已经成为表观基因组的精准医疗研究的首选方式。这种既定的抑制性染色质标记有多种检测选择:甲基化敏感的PCR和限制性酶、基于抗体的方法(如MeDIP)或亚硫酸氢盐转化后的测序(BS-seq/WGBS)36,37。DNA甲基化与诊断学的结合带来了令人兴奋的进展:
案例研究5:中枢神经系统(CNS)肿瘤的分类和治疗选择
许多原发性中枢神经系统肿瘤难以用传统的组织学方法进行分类38。例如,中枢神经系统神经外胚层肿瘤是类别广泛,其组织学特征是小的、分化不良的神经元和神经胶质。在一项研究中,DNA甲基化分析的整合将75%以上的肿瘤重新分类为更具体的亚型,如髓母细胞瘤、高级胶质瘤、室管膜瘤和松果体瘤39。在其他原发性中枢神经系统肿瘤中,如胶质母细胞瘤,选择性启动子(如MGMT)的甲基化促进治疗的灵敏度,如替莫唑胺(temozolomide),指导治疗选择40。
案例研究6:无创体外诊断的发展
在早期研究中,在患者血浆或血清中的循环无细胞核小体上能够检测到DNA甲基化,并在一些概念验证的临床研究中观察到,如肺癌、乳腺癌和结直肠癌37,41-43。这为开发FDA批准的体外诊断测试铺平了道路,这些测试来自容易获得的样本,如Cologuard®和Epi proColon®的粪便或血液,以及几种有前景的液体活检测试,包括GRAIL的Galleri™,以检测癌症,无论其类型如何44-46。
然而,由于无法提供不同染色质区间的细节,特别是组织/细胞特异性增强子的活性,DNA甲基化的研究对某些应用来说是不够的。此外,DNA甲基化并不总是一种抑制性标记47,这意味着它对基因表达的影响可能是模糊的。虽然DNA甲基化已经证明破译表观基因组可以为精准医疗方法提供信息,但就揭示表观基因组学和染色质上的基因表达修饰机制而言,这只是冰山一角。
组蛋白翻译后修饰
染色质上显著的化学和蛋白质多样性充满了潜在的精准医疗靶点。具体而言,组蛋白PTMs和染色质结合蛋白(如转录因子)对基因表达产生了强大而动态的影响。迄今为止,超过100种独特的组蛋白PTMs或其组合被认为与人类疾病有关,包括多种癌症48-53,与此同时人们也发现组蛋白PTM模式的整体改变可以预测疾病状态、复发情况和患者的治疗应答54-56。
与DNA甲基化甚至染色质可及性检测(如ATAC-seq)不同,组蛋白PTMs提供了对不同、特定基因组特征的认知。全基因组染色质图谱工具的开发有助于将组蛋白PTMs分配到特定的基因组区间(Figure 2),如活跃的增强子(H3K27ac)、活跃的启动子(H3K4me3)、活跃的基因体(H3K36me3),甚至被抑制的基因(H3K9me2/3,H3K27me3)。因此,研究组蛋白PTMs可以阐明基因调控格局的多样性,包括细胞的功能。一个典型的例子是二价H3K4me3/H3K27me3标记,它表示未分化的干细胞或原生细胞中 '准备好'激活的增强子和启动子57。值得注意的是,在癌症中经常观察到二价基因表达的改变58,它们可能与病情恶化59或耐药性60有关。
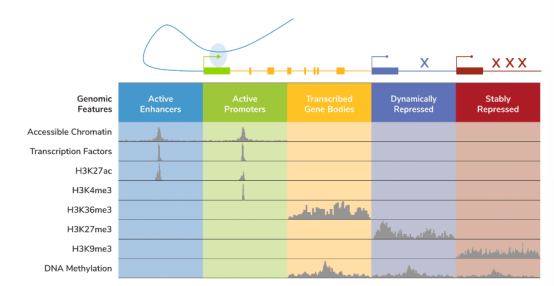
Figure 2 Overview of the localization of chromatin features.
因此,基因组图谱研究包含了个性化医疗的关键信息。这一领域的前沿研究已经在精准医疗领域产生了影响:
案例研究7:癌症治疗反应的预测和调节
一项研究发现,组蛋白PTM H3K27ac是增强子和活跃转录程序的标志物,可用于区分结直肠癌的亚类型,包括预后结果和治疗应答61。此外,使用这些增强子亚型分类的新型组合疗法在患者衍生的异种移植中显示出疗效。另一项研究发现,抑制性组蛋白PTM H3K27me3使乳腺癌细胞对化疗敏感,而阻止这一标记会抑制药物耐受性并延缓肿瘤形成60,这表明组蛋白PTMs具有治疗潜力。
这些研究共同表明,表观基因组研究具有指导临床决策和药物开发的强大潜力。
与整合表观基因组学有关的挑战
虽然染色质图谱展示了精准医疗应用的巨大前景,但表观基因组在临床应用方面的研究一直极具挑战性。
历史上,染色质图谱是通过ChIP-seq进行的。虽然具有突破性,但也有很大的局限性,这种局限阻碍了它在精准医疗中的应用。具体来说,这些限制包括:
● 低灵敏度和低通量,需要数以百万计的细胞,深度测序,以及大量的实践时间。
● 难以适应不同的细胞类型,需要对每个新的细胞类型进行优化。
● 组蛋白PTMs的非特异性抗体——事实上,绝大多数(如EpiCypher62所示,>70%)与相关标记发生交叉反应。
● 所有这些都将导致高成本和低质量的数据!
这些挑战共同限制了ChIP-seq数据的规模、应用和解读。ChIP的可靠性差,也使得它难以研究疾病的异质性和患者的特定治疗应答。
总之,ChIP-seq无法完成表观基因组学在精准医疗方面所需的工作。那么还有什么其他选择吗?
CUT&RUN和CUT&Tag:改进的精准医疗表观基因组学研究工具
CUT&RUN与CUT&Tag分析解决了许多与ChIP-seq相关的问题,只需取用少量细胞和测序读数就能生成高分辨率的图谱,并且十分快速——EpiCypher的CUTANA™ CUT&RUN与CUT&Tag分析法从细胞到测序文库只需2到3天,而ChIP需要一周(或更长时间)。
CUT&RUN和CUT&Tag是研究的理想选择,可以提高我们对疾病风险变异、基因调控程序和诊断/治疗生物标志物的认识。与ChIP-seq相比,CUT&RUN和CUT&Tag与个性化医学应用相关的优势和特点包括:
● 高灵敏度和高通量:经过简化的工作流程,检测只需要5,000个细胞
● 广泛的细胞类型兼容性:干细胞、免疫细胞、患者衍生的异种细胞、FACS分选的细胞以及小鼠和人类原代细胞、冷冻样品和轻度交联的材料
● 经验证的抗体,以确保您对结果的信赖
● 成本低——与ChIP-seq相比,每个反应大约减少70%的成本。
CUT&RUN对大多数靶标都很稳健,包括瞬时相互作用蛋白(如染色质重塑酶)、先驱因子、异染色质靶点,并具有单细胞兼容试验方案63,能分析异质组织。重要的是,CUT&Tag只推荐用于组蛋白PTM靶点。
许多课题组已经使用EpiCypher的CUT&RUN64-67和CUT&Tag68,69检测来研究染色质结构并用于生物医学研究。其他文献引用已经证明了这项技术在精准医疗方面的前景:
案例研究8:神经元中与疾病相关的增强子鉴定
通过结合H3K27ac CUT&RUN、ATAC-seq和RNA-seq,一组便能够识别兴奋性与抑制性人类神经元特有的增强子70。增强子图谱与精神疾病风险变异数据库相结合,显示出与精神分裂症、多动症和双相情感障碍有关的变异富集。这项研究直接证明了表观基因组学与现有数据集的整合可以进一步了解疾病风险变异体的功能和异常基因的表达,从而推动精准医疗。
案例研究9:液体活检转录印迹的鉴定
CUT&RUN已被用于识别异种移植中ER+乳腺癌的转录印迹71。转录图谱被用来对人类乳腺肿瘤进行亚分类,而且值得注意的是,在患者的血浆中也能检测到71。这些研究直接证明了二代CUT&RUN基因组图谱技术在推进新兴的无创液体活检个性化医学应用方面的潜力。
结论和展望
尽管精准医疗面临着定义患者特异性分子特征的困难,不可否认它是一个令人振奋的领域。用于精准医疗的染色质研究有可能在两个主要方面显著改变该领域:
1. 新型染色质生物标志物的鉴定和验证——在现有的基因组和转录组方法的基础上展现了一个需要监管的、环境敏感的尚未开发领域。
2. 阐明驱动疾病的转录调控机制——这将有助于表征疾病风险变异,并确定新的转录生物标志物和药物靶点。
CUT&RUN与CUT&Tag是能够用于实现这些目标的强大检测方法。表观基因组学在精准医疗中的其他应用包括精细绘制独特细胞类型中的染色质区室,以帮助阐明疾病发展和进展的染色质调节机制。这种表观基因组图谱可与其他测序方式相结合,包括DNA甲基化、RNA-seq、ATAC-seq或Hi-C,以表征个别患者的疾病特征并改善临床预后。
参考文献
1. Jørgensen JT. Twenty Years with Personalized Medicine: Past, Present, and Future of Individualized Pharmacotherapy. Oncologist 24, e432-e40 (2019). PubMed PMID: 30940745.
2. Ashley EA. Towards precision medicine. Nat Rev Genet 17, 507-22 (2016). PubMed PMID: 27528417.
3. Ikegawa S. A short history of the genome-wide association study: where we were and where we are going. Genomics Inform 10, 220-5 (2012). PubMed PMID: 23346033.
4. Uffelmann E et al. Genome-wide association studies. Nature Reviews Methods Primers 1, 59 (2021). https://doi.org/10.1038/s43586-021-00056-9.
5. Wang K et al. Diverse genome-wide association studies associate the IL12/IL23 pathway with Crohn Disease. Am J Hum Genet 84, 399-405 (2009). PubMed PMID: 19249008.
6. Moschen AR et al. IL-12, IL-23 and IL-17 in IBD: immunobiology and therapeutic targeting. Nat Rev Gastroenterol Hepatol 16, 185-96 (2019). PubMed PMID: 30478416.
7. Greulich H. The genomics of lung adenocarcinoma: opportunities for targeted therapies. Genes Cancer 1, 1200-10 (2010). PubMed PMID: 21779443.
8. Targeted Therapies for Lung Cancer: American Lung Association. Available from: https://www.lung.org/lung-health-diseases/lung-disease-lookup/lung-cancer/treatment/types-of-treatment/targeted-therapies.
9. Boyle MP et al. A CFTR corrector (lumacaftor) and a CFTR potentiator (ivacaftor) for treatment of patients with cystic fibrosis who have a phe508del CFTR mutation: a phase 2 randomised controlled trial. Lancet Respir Med 2, 527-38 (2014). PubMed PMID: 24973281.
10. Condren ME et al. Ivacaftor: a novel gene-based therapeutic approach for cystic fibrosis. J Pediatr Pharmacol Ther 18, 8-13 (2013). PubMed PMID: 23616732.
11. Wainwright CE et al. Lumacaftor-Ivacaftor in Patients with Cystic Fibrosis Homozygous for Phe508del CFTR. N Engl J Med 373, 1783-4 (2015). PubMed PMID: 26510034.
12. Edwards SL et al. Beyond GWASs: illuminating the dark road from association to function. Am J Hum Genet 93, 779-97 (2013). PubMed PMID: 24210251.
13. Gasperini M et al. Towards a comprehensive catalogue of validated and target-linked human enhancers. Nat Rev Genet 21, 292-310 (2020). PubMed PMID: 31988385.
14. Morley M et al. Genetic analysis of genome-wide variation in human gene expression. Nature 430, 743-7 (2004). PubMed PMID: 15269782.
15. Schadt EE et al. An integrative genomics approach to infer causal associations between gene expression and disease. Nat Genet 37, 710-7 (2005). PubMed PMID: 15965475.
16. Emilsson V et al. Genetics of gene expression and its effect on disease. Nature 452, 423-8 (2008). PubMed PMID: 18344981.
17. McGovern A et al. Capture Hi-C identifies a novel causal gene, IL20RA, in the pan-autoimmune genetic susceptibility region 6q23. Genome Biol 17, 212 (2016). PubMed PMID: 27799070.
18. Smemo S et al. Obesity-associated variants within FTO form long-range functional connections with IRX3. Nature 507, 371-5 (2014). PubMed PMID: 24646999.
19. Bradner JE et al. Transcriptional Addiction in Cancer. Cell 168, 629-43 (2017). PubMed PMID: 28187285.
20. Nagafuchi Y et al. Lessons From Transcriptome Analysis of Autoimmune Diseases. Front Immunol 13, 857269 (2022). PubMed PMID: 35663941.
21. Cooper-Knock J et al. Gene expression profiling in human neurodegenerative disease. Nat Rev Neurol 8, 518-30 (2012). PubMed PMID: 22890216.
22. Liu Y et al. RNA-Seq identifies novel myocardial gene expression signatures of heart failure. Genomics 105, 83-9 (2015). PubMed PMID: 25528681.
23. Lee TI et al. Transcriptional regulation and its misregulation in disease. Cell 152, 1237-51 (2013). PubMed PMID: 23498934.
24. Siegel RL et al. Cancer statistics, 2022. CA Cancer J Clin 72, 7-33 (2022). PubMed PMID: 35020204.
25. Cancer Stat Facts: Female Breast Cancer: National Cancer Institute. Available from: https://seer.cancer.gov/statfacts/html/breast.html.
26. Slamon DJ et al. Use of chemotherapy plus a monoclonal antibody against HER2 for metastatic breast cancer that overexpresses HER2. N Engl J Med 344, 783-92 (2001). PubMed PMID: 11248153.
27. Sotiriou C et al. Gene-expression signatures in breast cancer. N Engl J Med 360, 790-800 (2009). PubMed PMID: 19228622.
28. Zhao W et al. SCD5 expression correlates with prognosis and response to neoadjuvant chemotherapy in breast cancer. Sci Rep 11, 8976 (2021). PubMed PMID: 33903614.
29. Yuan Q et al. Identifying Differential Expression Genes and Prognostic Signature Based on Subventricular Zone Involved Glioblastoma. Front Genet 13, 912227 (2022). PubMed PMID: 35873494.
30. Knowles DA et al. Allele-specific expression reveals interactions between genetic variation and environment. Nat Methods 14, 699-702 (2017). PubMed PMID: 28530654.
31. Favé MJ et al. Gene-by-environment interactions in urban populations modulate risk phenotypes. Nat Commun 9, 827 (2018). PubMed PMID: 29511166.
32. Prescott S et al. The role of epigenetic dysregulation in the epidemic of allergic disease. Clin Epigenetics 2, 223-32 (2011). PubMed PMID: 21949548.
33. Heijmans BT et al. Persistent epigenetic differences associated with prenatal exposure to famine in humans. Proc Natl Acad Sci U S A 105, 17046-9 (2008). PubMed PMID: 18955703.
34. Breitling LP et al. Smoking, F2RL3 methylation, and prognosis in stable coronary heart disease. Eur Heart J 33, 2841-8 (2012). PubMed PMID: 22511653.
35. Sheffield NC et al. Identifying and characterizing regulatory sequences in the human genome with chromatin accessibility assays. Genes (Basel) 3, 651-70 (2012). PubMed PMID: 24705081.
36. Mattei AL et al. DNA methylation: a historical perspective. Trends Genet 38, 676-707 (2022). PubMed PMID: 35504755.
37. Laird PW. The power and the promise of DNA methylation markers. Nat Rev Cancer 3, 253-66 (2003). PubMed PMID: 12671664.
38. Galbraith K et al. DNA methylation as a diagnostic tool. Acta Neuropathol Commun 10, 71 (2022). PubMed PMID: 35527288.
39. Sturm D et al. New Brain Tumor Entities Emerge from Molecular Classification of CNS-PNETs. Cell 164, 1060-72 (2016). PubMed PMID: 26919435.
40. Butler M et al. MGMT Status as a Clinical Biomarker in Glioblastoma. Trends Cancer 6, 380-91 (2020). PubMed PMID: 32348734.
41. Li P et al. Liquid biopsies based on DNA methylation as biomarkers for the detection and prognosis of lung cancer. Clin Epigenetics 14, 118 (2022). PubMed PMID: 36153611.
42. Manoochehri M et al. DNA methylation biomarkers for noninvasive detection of triple-negative breast cancer using liquid biopsy. Int J Cancer 152, 1025-35 (2023). PubMed PMID: 36305646.
43. Jin S et al. Efficient detection and post-surgical monitoring of colon cancer with a multi-marker DNA methylation liquid biopsy. Proc Natl Acad Sci U S A 118, (2021). PubMed PMID: 33495330.
44. Pickhardt PJ. Emerging stool-based and blood-based non-invasive DNA tests for colorectal cancer screening: the importance of cancer prevention in addition to cancer detection. Abdom Radiol (NY) 41, 1441-4 (2016). PubMed PMID: 27259335.
45. Nadauld LD et al. The PATHFINDER Study: Assessment of the Implementation of an Investigational Multi-Cancer Early Detection Test into Clinical Practice. Cancers (Basel) 13, (2021). PubMed PMID: 34298717.
46. Taryma-Leśniak O et al. Current status of development of methylation biomarkers for in vitro diagnostic IVD applications. Clin Epigenetics 12, 100 (2020). PubMed PMID: 32631437.
47. Rauluseviciute I et al. DNA hypermethylation associated with upregulated gene expression in prostate cancer demonstrates the diversity of epigenetic regulation. BMC Med Genomics 13, 6 (2020). PubMed PMID: 31914996.
48. Chopra M et al. Disturbing the histone code in leukemia: translocations and mutations affecting histone methyl transferases. Cancer Genet 208, 192-205 (2015). PubMed PMID: 25592767.
49. Greenblatt SM et al. Chromatin modifiers and the promise of epigenetic therapy in acute leukemia. Leukemia 28, 1396-406 (2014). PubMed PMID: 24609046.
50. Gajer JM et al. Histone acetyltransferase inhibitors block neuroblastoma cell growth in vivo. Oncogenesis 4, e137 (2015). PubMed PMID: 25664930.
51. Witt O et al. Targeting histone deacetylases in neuroblastoma. Curr Pharm Des 15, 436-47 (2009). PubMed PMID: 19199971.
52. Hanmod SS et al. Targeting histone deacetylases (HDACs) and Wee1 for treating high-risk neuroblastoma. Pediatr Blood Cancer 62, 52-9 (2015). PubMed PMID: 25308916.
53. Kobayashi K et al. Epigenetic regulation of the neuroblastoma genes, Arid3b and Mycn. Oncogene 32, 2640-8 (2013). PubMed PMID: 22751132.
54. Seligson DB et al. Global histone modification patterns predict risk of prostate cancer recurrence. Nature 435, 1262-6 (2005). PubMed PMID: 15988529.
55. Kamińska K et al. Prognostic and Predictive Epigenetic Biomarkers in Oncology. Mol Diagn Ther 23, 83-95 (2019). PubMed PMID: 30523565.
56. Seligson DB et al. Global levels of histone modifications predict prognosis in different cancers. Am J Pathol 174, 1619-28 (2009). PubMed PMID: 19349354.
57. Macrae TA et al. Regulation, functions and transmission of bivalent chromatin during mammalian development. Nat Rev Mol Cell Biol 24, 6-26 (2023). PubMed PMID: 36028557.
58. Kumar D et al. Decoding the function of bivalent chromatin in development and cancer. Genome Res 31, 2170-84 (2021). PubMed PMID: 34667120.
59. Chaffer CL et al. Poised chromatin at the ZEB1 promoter enables breast cancer cell plasticity and enhances tumorigenicity. Cell 154, 61-74 (2013). PubMed PMID: 23827675.
60. Marsolier J et al. H3K27me3 conditions chemotolerance in triple-negative breast cancer. Nat Genet 54, 459-68 (2022). PubMed PMID: 35410383.
61. Orouji E et al. Chromatin state dynamics confers specific therapeutic strategies in enhancer subtypes of colorectal cancer. Gut 71, 938-49 (2022). PubMed PMID: 34059508.
62. Shah RN et al. Examining the Roles of H3K4 Methylation States with Systematically Characterized Antibodies. Mol Cell 72, 162-77.e7 (2018). PubMed PMID: 30244833.
63. Hainer SJ et al. Profiling of Pluripotency Factors in Single Cells and Early Embryos. Cell 177, 1319-29.e11 (2019). PubMed PMID: 30955888.
64. Janssens DH et al. Automated in situ chromatin profiling efficiently resolves cell types and gene regulatory programs. Epigenetics Chromatin 11, 74 (2018). PubMed PMID: 30577869.
65. Theisen ER et al. Chromatin profiling reveals relocalization of lysine-specific demethylase 1 by an oncogenic fusion protein. Epigenetics 16, 405-24 (2021). PubMed PMID: 32842875.
66. Garcia-Martinez L et al. Endocrine resistance and breast cancer plasticity are controlled by CoREST. Nat Struct Mol Biol 29, 1122-35 (2022). PubMed PMID: 36344844.
67. Dai X et al. Massively parallel knock-in engineering of human T cells. Nat Biotechnol (2023). PubMed PMID: 36702900.
68. Sparbier CE et al. Targeting Menin disrupts the KMT2A/B and polycomb balance to paradoxically activate bivalent genes. Nat Cell Biol 25, 258-72 (2023). PubMed PMID: 36635503.
69. Battistello E et al. Stepwise activities of mSWI/SNF family chromatin remodeling complexes direct T cell activation and exhaustion. Mol Cell 83, 1216-36.e12 (2023). PubMed PMID: 36944333.
70. Sanchez-Priego C et al. Mapping cis-regulatory elements in human neurons links psychiatric disease heritability and activity-regulated transcriptional programs. Cell Rep 39, 110877 (2022). PubMed PMID: 35649373.
71. Rao S et al. Transcription factor-nucleosome dynamics from plasma cfDNA identifies ER-driven states in breast cancer. Sci Adv 8, eabm4358 (2022). PubMed PMID: 36001652.

更多产品详情,请联系EpiCypher全国授权代理-欣博盛生物
全国服务热线: 4006-800-892 邮箱: market@neobioscience.com
深圳: 0755-26755892 北京: 010-88594029
广州:020-87615159 上海: 021-34613729
代理品牌网站: www.neobioscience.com
自主品牌网站: www.neobioscience.net
