磷酸化蛋白质组学简介
Levene和Alsberg在1906年首次发现了蛋白的磷酸化,这个PTM在近30年后被定位到蛋白的丝氨酸残基上(如图1)。由于四个领域的平行发展:(i)二维凝胶电泳(2D-PAGE);(ii)质谱方法;(iii)蛋白数据库;(iv)生物信息学工具,蛋白质组学研究在1990年代中期开始[1]。
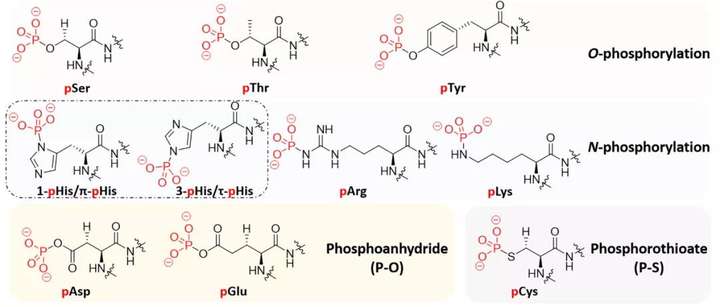
最常见的蛋白质磷酸化包括与丝氨酸、苏氨酸和酪氨酸的羟基侧链形成***键。两种拮抗酶系统(激酶和磷酸酶)分别催化蛋白质磷酸化和去磷酸化[2]。磷酸化被分为四类:O-phosphorylation(pSer, pThr, pTyr), N-phosphorylation(pHis, pArg, pLys), phosphoanhydride(pAsp, pGlu) and phosphorothioate (pCys)(见图2)[3]。
蛋白质磷酸化是许多细胞过程中的基本调节机制,磷酸化的异常扰动与各种人类疾病有关。因此,能够对磷酸化蛋白质组进行全系统定量分析将为揭示感兴趣的疾病的新信号通路、药物靶点和生物标志物提供强大的推动力。

数据分析
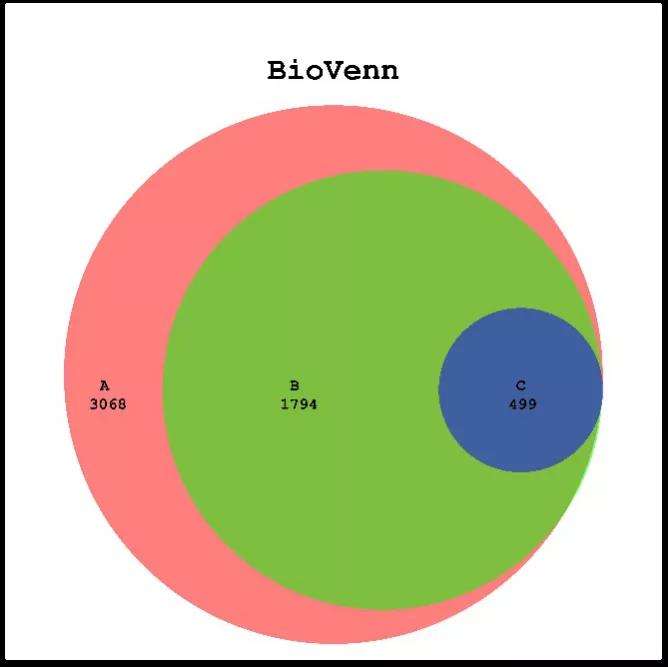
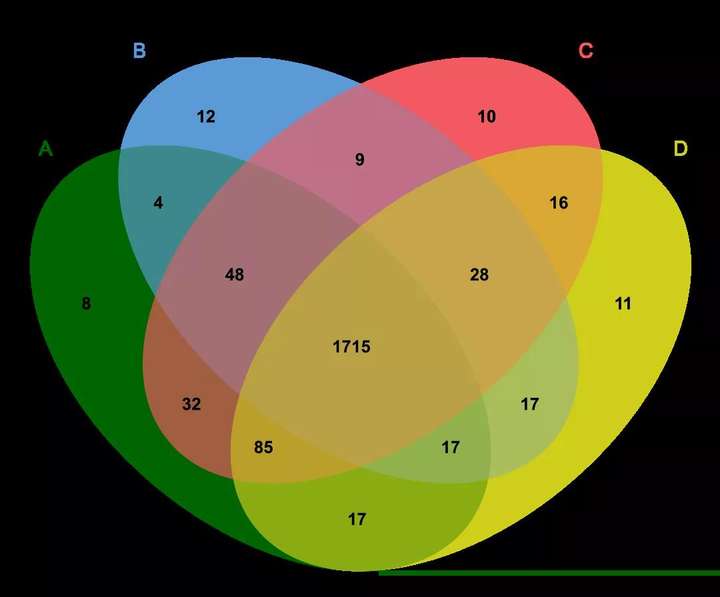
1.韦恩图
不同处理组之间的共性和差异通常可以用韦恩图展示[4]。BioVenn1能表现出不同组的元素数量以及相同点和不同点,同时展现数据集的大小。
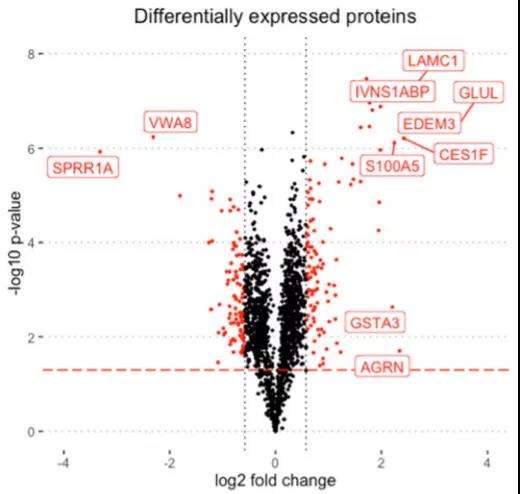
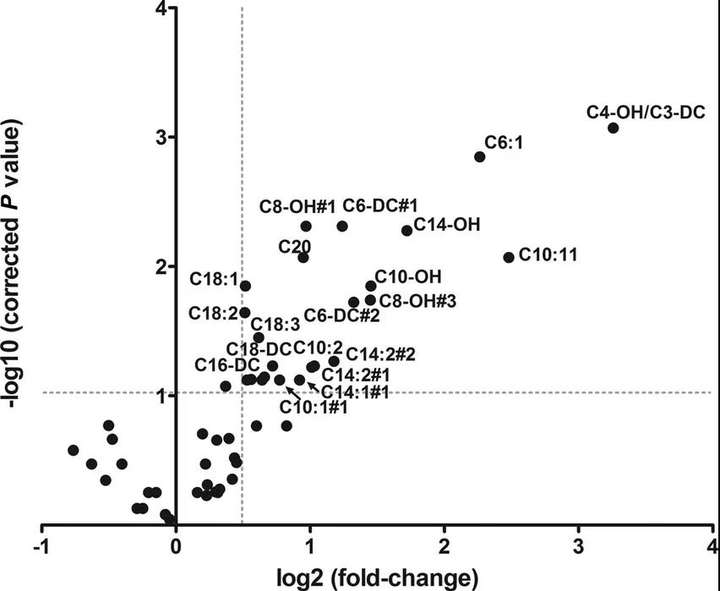
2.火山图
在CHO-K1细胞适应无谷氨酰胺培养基的生长的实验中,用火山图来表示组间差异表达蛋白的表达量变化,阈值设置为fold change < 1.5和p value < 0.05[5](如Volcano Plot-1)。并在图中标注TOP10差异表达蛋白。在实验中,表达量高且显著性高的差异蛋白是容易被关注的,并被选为目标蛋白。这也能降低后续的实验难度。
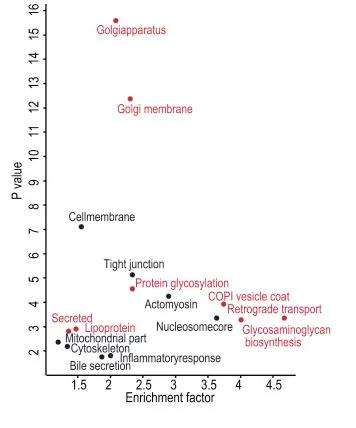
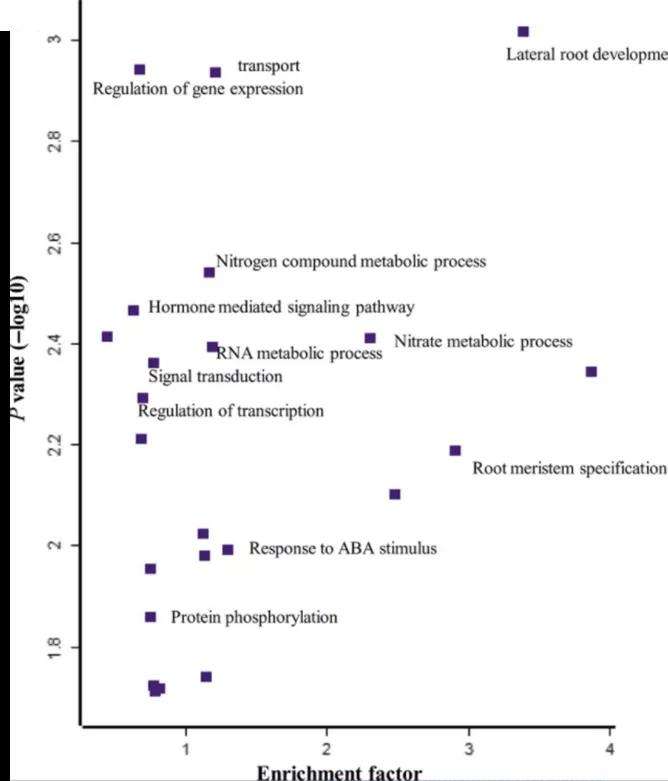
3.KEGG Pathway分析
磷酸化水平昼夜规律波动的肝脏蛋白显著富集在特定的代谢通路上,如胰岛素相关代谢途径、细胞自噬和昼夜节律相关的代谢途径(如图KEGG PATHWAY1-2),图中横坐标为富集程度Rich factor值,纵坐标表示矫正后的P-value值的大小,表明翻译后修饰在昼夜节律调控中起着关键作用。
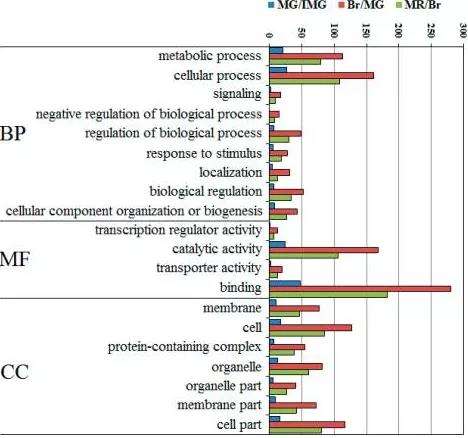
4.GO分析
GO分析旨在发掘与基因差异表达现象关联的单个特征基因功能类或多个特征功能类的组合。下面我们以辣椒的三个不同成长阶段为例,以直方图的形式展示了GO分析的结果。图中横坐标为映射差异表达蛋白的个数,纵坐标为GO Term。
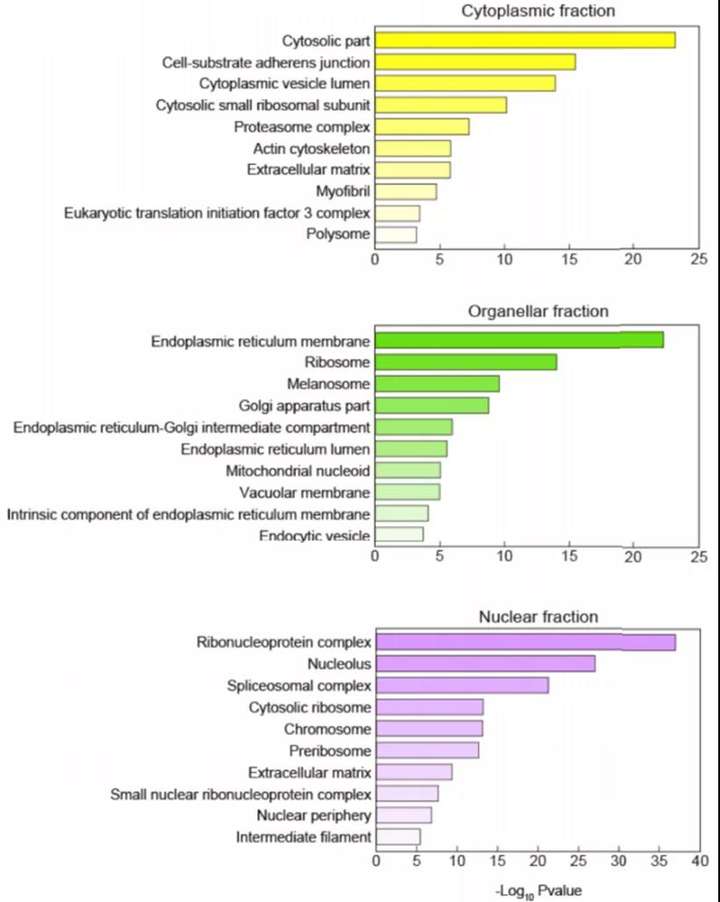
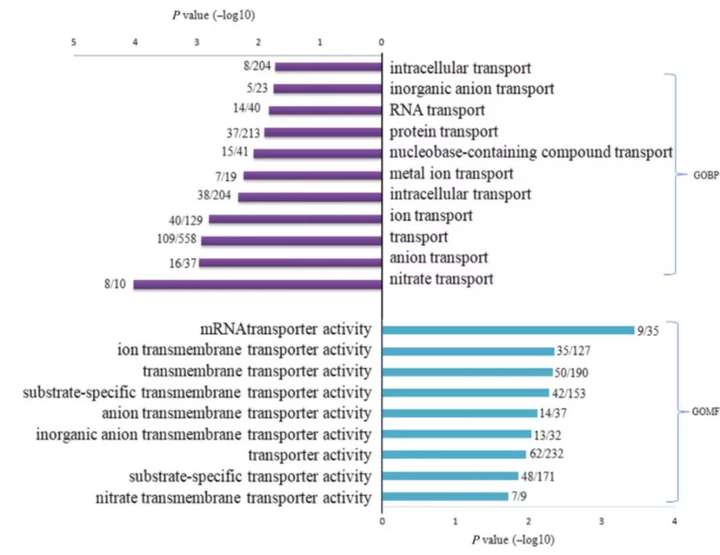
5.亚细胞定位
蛋白质必须转运到其应在的亚细胞结构上才能行使其功能,否则就会出现机体功能紊乱,产生各种疾病。蛋白质的位置是蛋白质最重要的属性之一,有助于确定蛋白质功能,揭示分子交互机理、理解复杂生理过程和开发药物靶标等方面的研究。
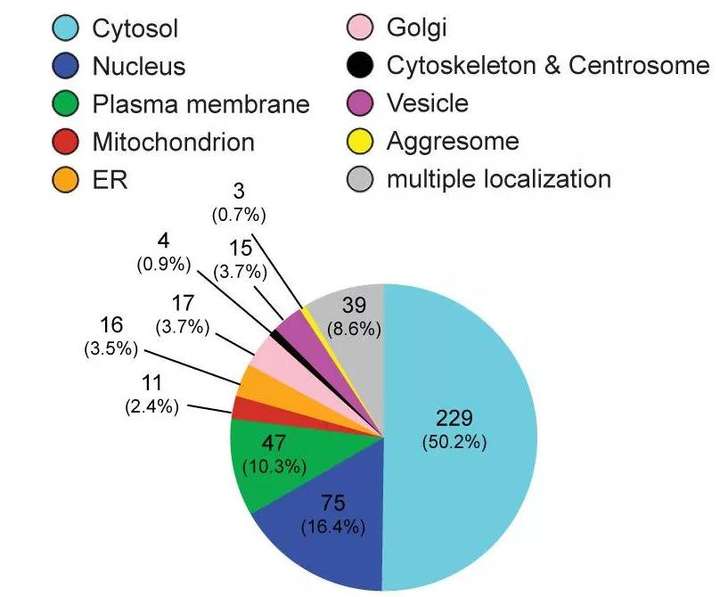
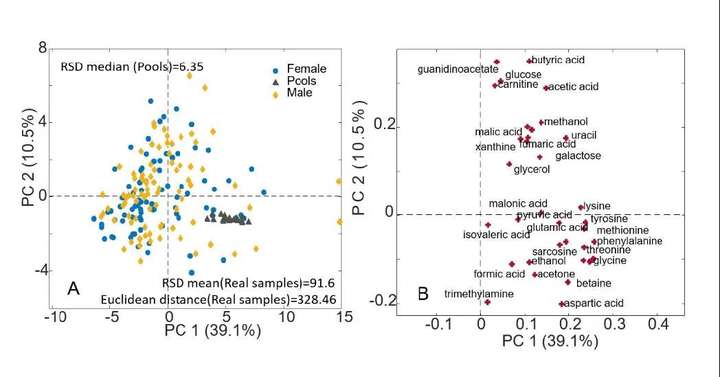
6.PCA分析
主成分分析 (PCA, principal component analysis)是一种数学降维方法, 利用正交变换 (orthogonal transformation)把一系列可能线性相关的变量转换为一组线性不相关的新变量(也称为主成分),从而利用新变量在更小的维度下展示研究对象的特征。用少数几个主成分的变化来近似代替原来多个变量的变化。通过主成分分析,可以反映不同样本间的关系;对于大量样本,通过离群点,判断是否存在离散样本,在进一步的分析中进行剔除。
7.PPI分析
蛋白质-蛋白质相互作用网络分析(Protein-Protein Interaction Network Analysis, PPI Network Analysis),是蛋白质组学的重要研究内容之一。蛋白质在行使生物功能时通过形成PPI网络以维持时间和空间上的协调一致,构建差异表达蛋白的相互作用网络,可以从蛋白质组层面发现差异表达蛋白的变化趋势,进一步帮助我们寻找差异表达蛋白中的关键节点。
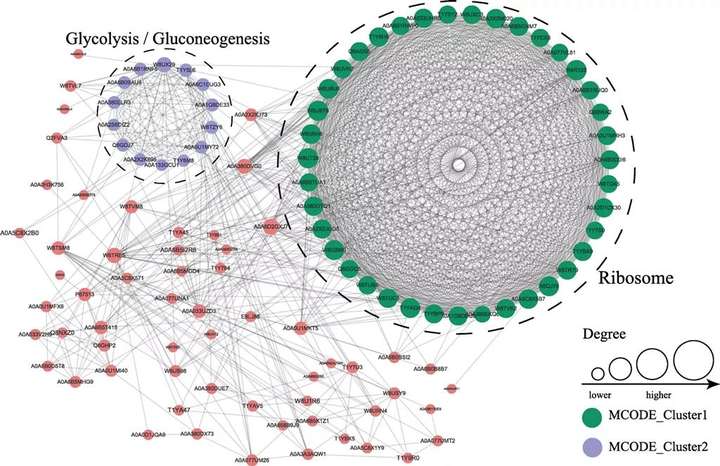
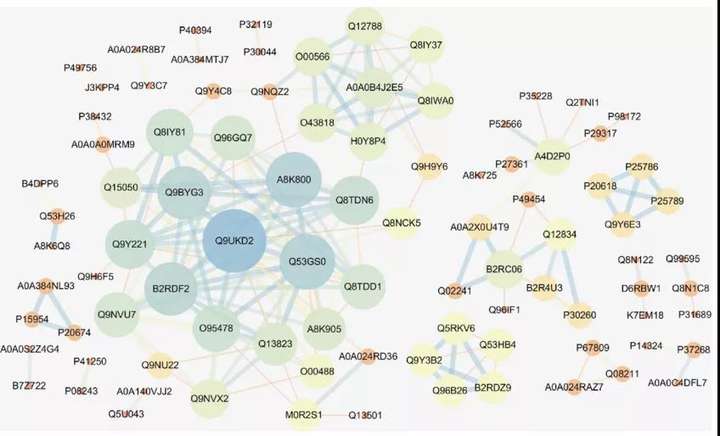
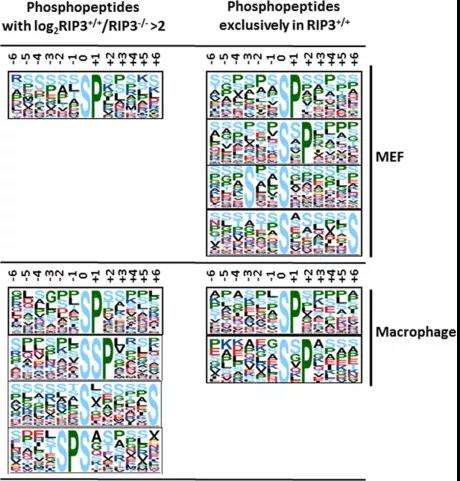
8.Motif分析
酶对特定底物的部分生化偏好可能是由修饰位点周围的残基决定的,这种蛋白或多肽序列形成的特定残基模式称为基序(Motif)在蛋白的同源序列中,不同位点的保守程度是不一样的,一般来说,对蛋白质功能和结构影响比较大的位点会比较保守,其它位点则不是很保守。这些保守的位点就称为“模体(motif)”。可以根据蛋白质序列特征(比如基于蛋白质基序)进行功能预测。

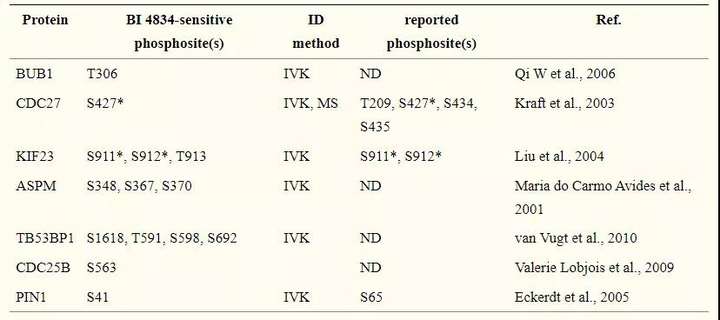
9.激酶(kinase)预测分析
蛋白激酶(protein kinases,简称PK)。催化蛋白质磷酸化过程的酶。蛋白质的磷酸化过程是神经信息在细胞内传递的最后环节,导致离子通道蛋白及通道门的状态变化。因此,激酶的变化与癌症和疾病密切相关。在磷酸化蛋白组分析中,通常利用motif来预测激酶,揭示参与分子过程的参与者。常用预测激酶网站:http://hprd.org/PhosphoMotif_finder。
参考文献
[1] Dupree, E.J., et al., A Critical Review of Bottom-Up Proteomics: The Good, the Bad, and the Future of this Field. Proteomes, 2020. 8(3).
[2] Kjeldsen, F., et al., On studying protein phosphorylation patterns using bottom-up LC-MS/MS: the case of human alpha-casein. Analyst, 2007. 132(8): p. 768-76.
[3] Huang, B., et al., NMR-based investigation into protein phosphorylation. Int J Biol Macromol, 2020. 145: p. 53-63.
[4] Panizza, E., et al., Isoelectric point-based fractionation by HiRIEF coupled to LC-MS allows for in-depth quantitative analysis of the phosphoproteome. Scientific Reports, 2017. 7(1): p. 4513.
[5] Kaushik, P., et al., LC-MS/MS-based quantitative proteomic and phosphoproteomic analysis of CHO-K1 cells adapted to growth in glutamine-free media. Biotechnology Letters, 2020. 42(12): p. 2523-2536.
[6] Ruiz M, Labarthe F, Fortier A, Bouchard B, Thompson Legault J, Bolduc V, Rigal O, Chen J, Ducharme A, Crawford PA, Tardif JC, Des Rosiers C. Circulating acylcarnitine profile in human heart failure: a surrogate of fatty acid metabolic dysregulation in mitochondria and beyond. Am J Physiol Heart Circ Physiol. 2017 Oct 1, 313(4).
[7] Krahmer N, Najafi B, Schueder F, et al. Uhlenhaut NH, Walther TC, Jungmann R, Zeigerer A, Borner GHH, Mann M. Organellar Proteomics and Phospho-Proteomics Reveal Subcellular Reorganization in Diet-Induced Hepatic Steatosis. Dev Cell. 2018 Oct 22;47(2): 205-221.
[8] Chen Y, Hoehenwarter W. Rapid and reproducible phosphopeptide enrichment by tandem metal oxide affinity chromatography: application to boron deficiency induced phosphoproteomics. Plant J. 2019 Apr;98(2):370-384.
[9] Liu Z, Lv J, Liu Y, Wang J, et al . Comprehensive Phosphoproteomic Analysis of Pepper Fruit Development Provides Insight into Plant Signaling Transduction. Int J Mol Sci. 2020 Mar 13;21(6).
[10] Masuda T, Sugiyama N, Tomita M, Ohtsuki S, Ishihama Y. Mass Spectrometry-Compatible Subcellular Fractionation for Proteomics. J Proteome Res. 2020 Jan 3;19(1):75-84.
[11] Zhang H, Cao X, Tang M, Zhong G, Si Y, Li H, Zhu F, Liao Q, Li L, Zhao J, Feng J, Li S, Wang C, Kaulich M, Wang F, Chen L, et al. A subcellular map of the human kinome. Elife. 2021 May 14;10:e64943.
[12] Cui Mengni,Trimigno Alessia,Aru Violetta,Rasmussen Morten A,Khakimov Bekzod,Engelsen Søren Balling. Influence of Age, Sex, and Diet on the Human Fecal Metabolome Investigated by 1H NMR Spectroscopy.[J]. Journal of proteome research,2021.
[13] Shi Y , Zhu J , Xu Y , et al. Malonyl-proteome profiles of Staphylococcus aureus reveal lysine malonylation modification in enzymes involved in energy metabolism[J]. Proteome Science, 2021,19(1).
[14] Li H , Y Zhang, Zhang J , et al. A quantitative proteomics analysis for small molecule Stemazole’s effect on human neural stem cells[J]. Proteome Science, 2020, 18(1).
[15] Wu X, Tian L, Li J, et al. Investigation of receptor interacting protein (RIP3)-dependent protein phosphorylation by quantitative phosphoproteomics. Mol Cell Proteomics. 2012 Dec;11(12):1640-51.
[16] Grosstessner-Hain K, Hegemann B, Novatchkova M, et al. Quantitative phospho-proteomics to investigate the polo-like kinase 1-dependent phospho-proteome. Mol Cell Proteomics. 2011, 10(11).
